Les vaccins Covid-19 à ARNm contiennent des quantités excessives d’ADN bactérien : preuves et implications
Des études récentes menées par Kevin McKernan, expert en méthodes de séquençage de l’ADN et de l’ARN, ont révélé que les lots de vaccins à ARNm modifié produits par Pfizer et Moderna contiennent une forte proportion d’ADN bactérien contaminant. Au total, l’ADN représente jusqu’à 20 à 35 % des acides nucléiques contenus dans chacun des lots de vaccins. Ces concentrations alarmantes dépassent de loin les niveaux considérés comme sûrs par des organismes de normalisation tels que l’Agence européenne des médicaments (EMA). Ce document résume les preuves de cette contamination par l’ADN et examine les risques possibles pour la santé des personnes vaccinées.
1. Le rôle de l’ADN dans la fabrication des vaccins ARNm
1.1. Contexte général
La plupart des lecteurs savent que
- les ARN synthétiques contenus dans les vaccins ARNm COVID-19 codent pour la protéine de pointe du SARS-CoV-2 ;
- dans les cellules vivantes de mammifères, les instructions pour construire une molécule de protéine donnée sont stockées sous la forme d’un gène dans l’ADN à l’intérieur du noyau ;
- pour construire une molécule de protéine donnée, la cellule transcrit d’abord son gène en ARN et modifie les deux extrémités de cette molécule pour former l’ARN messager (ARNm). L’ARNm est ensuite transporté du noyau vers le cytoplasme, où il incite les usines à protéines de la cellule – les ribosomes – à traduire la séquence nucléotidique de l’ARNm en la séquence d’acides aminés correspondante et à assembler la protéine.
1.2. étapes de la fabrication d’un vaccin à base d’ARNm
La protéine de Spike étant une grosse molécule, l’ARNm qui la code l’est également. La synthèse chimique totale de grandes molécules d’ARNm n’est pas pratique à grande échelle. Par conséquent, pour obtenir la molécule d’ARNm codant pour la protéine spike, le processus par lequel les cellules produisent leurs propres ARNm est imité in vitro. Ce processus comprend les étapes suivantes :
- Une copie d’ADN du gène de la protéine spike est insérée dans un plasmide bactérien. Il s’agit d’une molécule d’ADN double brin en forme d’anneau qui peut exister dans une cellule bactérienne indépendamment de l’ADN chromosomique de la cellule et qui peut également être copiée et transmise aux deux cellules filles lors de la division de la cellule.
- Le plasmide recombinant (artificiel) portant le gène de la protéine spike est introduit dans une cellule de l’espèce bactérienne Escherichia coli (E. coli ). Comme les cellules d’E.coli se divisent très rapidement, cette cellule unique peut, en peu de temps, se transformer en un très grand nombre de cellules. Chacune de ces cellules progénitrices contiendra ses propres copies héritées du plasmide et donc du gène de la protéine spike. Bien qu’il y ait une certaine probabilité que le plasmide soit perdu par certains des descendants au cours des divisions cellulaires successives, nous pouvons assurer son maintien en lui donnant un marqueur sélectionnable, qui garantit que seules les cellules qui conservent le plasmide survivront. Dans le cas des plasmides utilisés par Pfizer et Moderna, ce marqueur sélectionnable est un gène qui confère aux cellules hôtes une résistance à l’antibiotique kanamycine. Pour appliquer la sélection, les bactéries sont simplement cultivées en présence de kanamycine.
- Après avoir cultivé un nombre suffisant de cellules bactériennes dans un bouillon nutritif contenant de la kanamycine, ces cellules sont décomposées et l’ADN plasmidique est purifié des autres composants des cellules bactériennes.
- Les molécules de plasmide en forme d’anneau sont converties en forme linéaire à l’aide d’une enzyme de restriction, qui clive les deux brins de la molécule d’ADN à un site spécifique et unique situé en aval du gène de la protéine spike. Cette étape est nécessaire pour éviter la formation de molécules d’ARN trop longues qui peuvent avoir des effets indésirables in vivo. Les molécules d’ADN linéarisées peuvent être purifiées des molécules circulaires restantes, mais la manière et l’efficacité de cette opération dans la production des vaccins Pfizer et Modern ne sont pas connues du public.
- Une ARN polymérase est utilisée, en présence des nucléosides et des cofacteurs nécessaires, pour copier le gène de la protéine spike de la version ADN sur le plasmide linéarisé à la version ARNm. Pfizer et Moderna utilisent tous deux l’ARN polymérase T7, dérivée du bactériophage éponyme. Cette enzyme se lie à une séquence promotrice apparentée, également dérivée de T7, qui a été incorporée dans le plasmide en amont du gène de la protéine spike. Cette interaction entre la polymérase et le promoteur initie la transcription. À ce stade, le nucléoside synthétique N-méthyl-pseudouridine (mψU) est incorporé dans l’ARN artificiel à la place du nucléoside uridine naturel. Lorsqu’il est administré sous forme de vaccin, l’ARN ainsi modifié stimule moins le système immunitaire inné que l’ARN contenant l’uridine naturelle. Il se traduit également plus efficacement en protéines et, dans certaines conditions, résiste mieux à la dégradation[1]. Les vaccins ARNm de Pfizer et de Moderna contiennent du mψU au lieu de l’uridine naturelle.
- Les deux extrémités de chaque molécule d’ARN sont couplées par voie enzymatique à certaines parties qui se trouvent également à ces positions dans les ARNm naturels des mammifères et qui renforcent leur activité biologique et leur stabilité in vivo.
Ces étapes permettent d’obtenir un ARNm fonctionnel capable d’ordonner aux ribosomes des cellules de produire la protéine spike. Toutefois, à ce stade, le produit n’est pas encore pur : tout l’ADN matrice d’origine bactérienne est encore présent. Ce dernier ne doit pas être inclus dans le produit pharmaceutique final, car il présente des risques pour la santé des receveurs (voir section 4). Pour se débarrasser de cet ADN, une autre enzyme appelée DNase est ajoutée. Celle-ci devrait décomposer l’ADN en fragments plus petits, qui peuvent ensuite être séparés des molécules d’ARN beaucoup plus grandes par filtration et autres techniques de purification. Dans la dernière étape, l’ARNm est combiné à un mélange de lipides afin de le conditionner dans des nanoparticules lipidiques (LNP), qui incitent les cellules humaines à absorber la molécule d’ARNm et à fabriquer la protéine spike.
2. Que savions-nous auparavant sur le problème de la contamination par l’ADN ?
En bref, très peu de choses. Les rapports d’évaluation de la FDA sur les deux vaccins[2,3] ne mentionnent pas du tout ce problème. Le rapport d’évaluation de l’Agence européenne des médicaments (EMA) sur le vaccin de Pfizer mentionne que « la robustesse de l’étape de digestion par la DNase n’est pas considérée comme ayant été démontrée de manière approfondie »[4, p. 17]. Des termes similaires sont utilisés dans le rapport de l’EMA sur le vaccin Moderna[5, p. 19f]. Cependant, sur la base de ces seules informations, il est impossible de dire si le problème a été considéré comme grave et quelles mesures correctives ont été exigées par l’organisme de réglementation, le cas échéant.
3. Preuves indépendantes de la contamination des produits ARNm par l’ADN
Le 3 avril 2023, Kevin McKernan a décrit ses récentes découvertes dans trois articles sur son site web Subtack[6-8]. Les expériences décrites dans les deux premiers rapports ont été réalisées sur des échantillons de vaccins « bivalents » récemment introduits par Pfizer et Moderna. Ces préparations ressemblent aux précédentes « monovalentes » par leur composition chimique, c’est-à-dire qu’elles devraient contenir de l’ARNm très pur, complexé avec un mélange de molécules lipidiques (semblables à des graisses) dans des nanoparticules ARNm/lipides. La seule différence entre les deux variétés est que les vaccins bivalents contiennent un mélange de deux ARNm codant pour deux variantes antigéniques de la protéine de Spike. Cela n’a aucune incidence sur le problème technique de la contamination par l’ADN en tant que tel. Nous notons toutefois que l’ampleur de la contamination par l’ADN peut varier d’un lot de production à l’autre et que, jusqu’à présent, seul un petit nombre de lots ont été caractérisés à cet égard.
3.1. Le premier rapport de McKernan
Dans une première étude[6], McKernan a caractérisé à la fois l’ARN et l’ADN contenus dans les vaccins à ARNm.
3.1.1 Extraction et caractérisation directe des acides nucléiques des vaccins
La première étape a consisté à éliminer les lipides afin d’obtenir les acides nucléiques purs. La méthode à base de solvant utilisée ne fait pas de distinction entre l’ADN et l’ARN – si les deux sont présents, ils seront tous les deux récupérés. Les acides nucléiques extraits ont été séparés en fonction de leur taille. Cela a révélé non seulement les espèces d’ARNm régulières et à pic complet attendues, mais aussi des fragments plus petits, qui avaient été notés précédemment par les régulateurs et dans des travaux publiés par l’un des fabricants[9]. De manière plus surprenante, des espèces d’ARN plus grandes que l’ARNm complet ont également été trouvées. Ces espèces n’ont pas encore été caractérisées.
3.1.2 Amplification des acides nucléiques extraits
Comme étape préparatoire à la détermination des séquences exactes de nucléotides des acides nucléiques extraits, ceux-ci ont été amplifiés par des méthodes PCR. Dans le cas de l’ARN, la PCR a été précédée d’une transcription inverse en ADN à l’aide d’une enzyme spécifique (transcriptase inverse). L’objectif principal de cette étude étant d’étudier l’ARN et non l’ADN, cette étape d’amplification a été biaisée contre l’ADN par l’ajout d’actinomycine D qui, dans les conditions expérimentales données, inhibe sélectivement la synthèse de l’ADN. Ainsi, des quantités relativement faibles d’ADN ont été récupérées dans l’échantillon amplifié. Toutefois, dans le cas du vaccin Pfizer, la quantité d’ADN déterminée dépassait déjà la limite fixée arbitrairement par l’EMA pour le rapport maximal admissible entre l’ADN et l’ARN.
3.1.3 Résultats du séquençage de l’ADN
Avec les produits Pfizer et Moderna, des séquences d’ADN de plasmides complets ont été obtenues, bien qu’une certaine ambiguïté subsiste dans le cas des plasmides Moderna. Les caractéristiques des séquences plasmidiques seront donc discutées en relation avec la deuxième étude de McKernan, qui a utilisé un ADN de plus en plus pur pour le séquençage et a donc fourni des résultats plus fiables.
3.2. Le deuxième rapport de McKernan
La deuxième étude[7] s’est attachée à quantifier et à caractériser la contamination par l’ADN qui avait été détectée qualitativement dans la première.
3.2.1. L’ADN plasmidique contenu dans les vaccins ARNm est capable de se propager dans les cellules bactériennes
Dans la première expérience, il a été déterminé si l’ADN plasmidique dont la présence avait été déduite des résultats des séquences précédentes est effectivement biologiquement fonctionnel, c’est-à-dire qu’il peut être introduit et persister dans les cellules bactériennes. À cette fin, des acides nucléiques ont de nouveau été extraits des échantillons de vaccin. Ces acides nucléiques ont été mélangés à une suspension de cellules d’E. coli qui avaient été rendues compétentes pour l’absorption d’ADN.
Après avoir incité ces cellules à absorber l’ADN et leur avoir laissé le temps de se rétablir, elles ont été réparties dans des boîtes de Pétri remplies d’un milieu de croissance solidifié contenant de la kanamycine. Comme indiqué précédemment, la kanamycine tue toute cellule d’E. coli qui ne contient pas de gène de résistance. Par conséquent, la croissance observée de colonies bactériennes dans ces boîtes de Petri a confirmé que certaines cellules avaient effectivement acquis une résistance à la kanamycine en absorbant et en propageant les plasmides. Ce phénomène a été observé à la fois avec les échantillons des vaccins Pfizer et Moderna.
Dans ce contexte, il convient de noter que seules les molécules plasmidiques circulaires, et non linéarisées, peuvent être introduites efficacement dans les cellules bactériennes. Le succès de cette expérience suggère donc qu’une partie des molécules de plasmide a échappé à l’étape de linéarisation (étape 4 de la section 1.2) et a traversé l’ensemble du processus de production sous la forme circulaire qui existe dans les cellules bactériennes. D’autre part, étant donné que le nombre de colonies bactériennes observées dans cette expérience n’était pas élevé, il est probable que la majeure partie de l’ADN avait effectivement été linéarisée. Étant donné que les risques biologiques liés à la présence d’ADN étranger dans notre propre corps peuvent varier selon qu’il s’agit d’ADN linéaire ou circulaire, la présence probable des deux formes dans les vaccins mérite d’être soulignée. Les proportions exactes d’ADN circulaire et linéaire dans les mélanges restent à déterminer.
3.2.2 L’abondance de l’ADN contaminant
Le deuxième résultat majeur de cette étude est la quantification par PCR de l’ADN et de l’ARNm contenus dans les échantillons de vaccins. Comme vous le savez peut-être, dans une réaction PCR, un segment choisi d’une séquence d’acide nucléique est réduit par synthèse enzymatique au cours de plusieurs cycles de réaction successifs. À partir du nombre de cycles (ou duplications) nécessaires pour atteindre une certaine seuil de concentration, on peut calculer combien de copies de la séquence cible étaient présentes au départ.
Dans ces expériences, le format expérimental choisi était la PCR multiplex, c’est-à-dire que deux séquences cibles ont été amplifiées dans un seul mélange réactionnel. L’une de ces cibles se trouvait dans le gène de la protéine spike et devait donc être présente à la fois dans les molécules d’ADN plasmidiques et dans les molécules d’ARNm de spike transcrites à partir de ces dernières. Afin d’inclure les molécules d’ARNm dans cette amplification, la PCR a de nouveau été précédée d’une transcription inverse.
L’autre séquence cible se trouvait dans le gène de résistance à la kanamycine, qui ne devrait être présent que dans l’ADN plasmidique. En comparant le nombre de cycles nécessaires pour que chacune des deux cibles franchisse le seuil, il a été déterminé que jusqu’à 35 % de l’acide nucléique total contenu dans les vaccins est en fait de l’ADN. À titre de comparaison, l’EMA a stipulé que l’ADN ne devait pas représenter plus de 0,033 % de l’ensemble des acides nucléiques.
3.2.3 Détermination des séquences d’ADN plasmidique
Les plasmides initialement contenus dans les vaccins et introduits dans des cellules bactériennes (voir section 3.2.1) ont été à nouveau isolés de ces cultures bactériennes et leurs séquences d’ADN complètes ont été déterminées. Ces séquences ont été fournies intégralement dans la première étude de McKernan[6], mais ce dernier a indiqué qu’il travaillait encore à corroborer et à affiner les données de séquençage. Entre-temps, les caractéristiques fonctionnelles de l’ADN plasmidique trouvé dans les échantillons de vaccins Pfizer sont présentées dans la figure 1. Elles seront examinées dans le cadre de l’évaluation des risques.
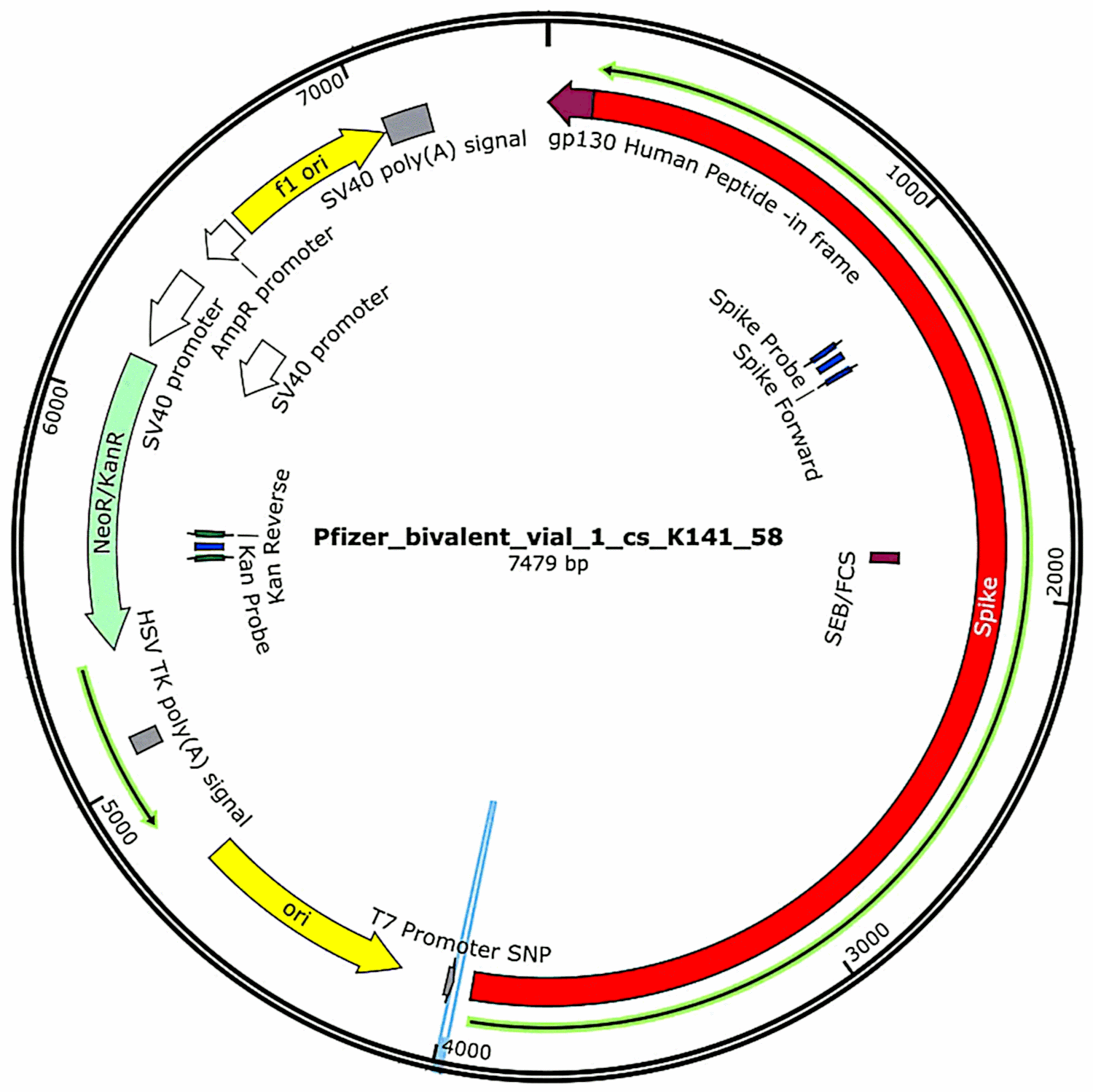
3.3. Le troisième rapport de McKernan
Dans son dernier rapport en date, McKernan a examiné huit flacons d’un lot antérieur du vaccin de Pfizer en utilisant la méthode de PCR quantitative décrite ci-dessus. Dans ce cas, la teneur en ADN était nettement inférieure à celle des échantillons de vaccins bivalents, mais dépassait encore la limite MPE (Maximum Permissible Exposure – Exposition Maximum Tolérée) d’un facteur de 18 à 70[8].
4. Évaluation des risques
Nous devons partir du principe que l’ADN recombinant contenu dans les vaccins ARNm peut être introduit dans les cellules de notre corps, et que cela sera facilité par les nanoparticules lipidiques, tout comme pour l’ARNm lui-même. Cela présente plusieurs types de risques pour la santé.
4.1. prolongation de la durée d’expression de la protéine spike
Un argument clé régulièrement utilisé pour promouvoir la sécurité perçue du vaccin à ARNm est que l’ARNm a une courte durée de vie in vivo et que l’expression de l’antigène codé sera donc également de courte durée. Par exemple, le rapport d’évaluation de l’EMA sur le vaccin Pfizer indique que des expériences animales avec un vaccin modèle ont été acceptées au lieu d’études adéquates sur le vaccin COVID-19 lui-même[4, p. 46] :
Comme prévu avec un produit ARNm, l’expression de la luciférase était transitoire … Le signal a lentement diminué au cours des 72 premières heures et après 6 et 9 jours, les signaux se sont encore affaiblis pour atteindre des niveaux environ 18 et 7 fois plus élevés que les signaux obtenus chez les animaux ayant reçu une injection de contrôle tamponnée.
Ces résultats semblent concorder avec deux études in vitro qui ont comparé la durée de l’expression protéique entre des espèces d’ARN messager dont la séquence était identique mais qui contenaient de l’uridine ou du mψU, respectivement ; comme indiqué précédemment, ce dernier est également présent dans les vaccins ARNm de Pfizer et de Moderna. Dans les deux études[1,10], les espèces d’ARN modifiées par le mψU ont induit des niveaux d’expression protéique significativement plus élevés, mais cette expression élevée a diminué avec une demi-vie (temps de diminuer de moitié sa concentration) similaire à celle de l’ARN non modifié. Aucune des demi-vies qui peuvent être déduites des données de l’une ou l’autre étude ne dépasse 4,5 jours.
Il ressort toutefois clairement de plusieurs études menées sur des personnes vaccinées que la protéine spike elle-même et les acides nucléiques qui la codent peuvent être détectés dans la circulation sanguine et dans divers organes pendant des semaines, voire des mois, après l’injection[11-15]. Cette divergence entre les études in vitro et in vivo a été jusqu’à présent difficile à comprendre. Les niveaux élevés d’ADN plasmidique résiduel dans les vaccins détectés par McKernan suggèrent maintenant une explication plausible.
Pour que l’ADN plasmidique bactérien permette l’expression prolongée de la protéine de pointe, deux conditions doivent être remplies :
- l’ADN plasmidique doit persister dans les cellules de notre corps, et
- le gène de la protéine spike de ce plasmide doit être transcrit en ARNm par notre propre ARN polymérase II cellulaire.
Bien que nous ne disposions pas encore de données expérimentales directes sur les plasmides d’expression du spike de Pfizer et Moderna, les précédents suggèrent qu’en fait, les deux conditions sont remplies. On a constaté que les plasmides recombinants exprimant le facteur IX de coagulation persistaient dans les cellules hépatiques d’animaux de laboratoire à des niveaux stables pendant une période allant jusqu’à un an et demi[16,17], ce qui correspondait à la durée totale de l’expérience. On pourrait objecter que les plasmides utilisés dans ces études étaient circulaires, alors que la plupart de l’ADN plasmidique contenu dans les vaccins ARNm est probablement sous forme linéaire (voir section 1.2). Nous répondons que, premièrement, une partie de l’ADN plasmidique circulaire subsiste probablement (voir section 3.2.1) et que, deuxièmement, il a été démontré que l’ADN viral recombinant persiste sous forme linéaire chez les animaux pendant des périodes de temps tout aussi longues[18], ce qui suggère qu’il pourrait en être de même pour l’ADN plasmidique.
Dans les études citées[16,17], le gène codant pour la protéine en question (facteur IX de coagulation) avait été placé sous le contrôle d’un promoteur mammifère, et la protéine du facteur IX a effectivement été exprimée à des niveaux stables pendant toute la durée de l’étude. En revanche, le gène de la protéine spike contenu dans les plasmides d’expression Pfizer et Moderna est sous le contrôle d’un promoteur de bactériophage T7. Nous ne pouvons pas supposer a priori que ce promoteur fonctionnera en l’absence de l’ARN polymérase T7 qu’il connaît. Il a toutefois été confirmé expérimentalement que le promoteur T7 se lie également à l’ARN polymérase II cellulaire et provoque l’expression de protéines dans les cellules de mammifères[19].
En résumé, il convient de prendre au sérieux la possibilité que l’expression durable observée de la protéine spike soit due à l’ADN plasmidique contenu dans les vaccins ARNm. La persistance prolongée de l’ARNm de la protéine spike et son expression après la vaccination, détectées dans les biopsies et les autopsies, ont été associées sans équivoque à des lésions graves[14,20], qui sont très probablement dues à une attaque immunitaire contre les cellules exprimant cet antigène étranger. L’omission d’études expérimentales correspondantes dans la phase d’essai préclinique, associée à l’ampleur de cette contamination, crée un risque de sécurité totalement inacceptable.
4.2. Risques liés aux séquences d’ADN réglementaires dérivées du SV40
Une caractéristique identifiée par McKernan dans les plasmides Pfizer mais pas dans les plasmides d’expression Moderna[6] est un promoteur dérivé du virus SV40, qui appartient à la famille des polyomes (voir section 4.2). Ce promoteur est situé en amont du gène de résistance à la kanamycine ; et comme il est actif dans les cellules de mammifères, la protéine codée par ce gène de résistance sera exprimée dans toute cellule contenant cet ADN. Comme la protéine spike, cette protéine est un antigène étranger et peut donc elle aussi déclencher une attaque immunitaire sur les cellules qui l’expriment.
Le promoteur du SV40 comprend également une origine de réplication interne qui peut potentiellement provoquer des copies du plasmide dans les cellules de mammifères[21]. Cela nécessite la présence du grand antigène viral T, une protéine qui reconnaît directement cette origine et initie alors la réplication de la molécule d’ADN. Cette protéine n’est pas codée par le plasmide et n’est pas non plus normalement présente dans les cellules de notre corps, mais peut être fournie soit par le virus SV40 lui-même, soit par un virus polyome apparenté. Une minorité de la population humaine est infectée de manière latente par le SV40, et cette infection latente est associée à certaines maladies malignes et non malignes[22]. Si une copie du plasmide Pfizer est introduite dans une cellule qui héberge le SV40, des copies supplémentaires de ce plasmide peuvent être formées.
Deux polyomavirus apparentés, beaucoup plus répandus dans la population humaine, sont le virus BK et le virus JC[23,24]. L’antigène JC large T est apparemment moins efficace en conjonction avec l’origine SV40 que la protéine SV40 elle-même[25], mais la réplication du plasmide Pfizer dans des cellules infectées de manière latente par les virus JC ou BK ne peut cependant pas être exclue. Des copies supplémentaires du plasmide générées de cette manière amplifieraient tous les autres risques discutés dans cette section, à l’exception peut-être de l’inflammation non spécifique (voir section 4.4).
4.3. Insertion génomique de l’ADN plasmidique
Les scénarios examinés jusqu’à présent impliquent tous la persistance épisodique et indépendante de l’ADN plasmidique ; celui-ci sera présent à proximité des chromosomes (dans le noyau cellulaire), mais ne sera devenu partie intégrante d’aucun d’entre eux. Ces molécules plasmidiques indépendantes et non réplicatives ont tendance à être perdues lors de la division cellulaire[26]. Cependant, comme nous le verrons, dans certains cas, une molécule de plasmide peut être intégrée dans l’un des chromosomes de sa cellule hôte, et sera alors héritée par tous les descendants de cette cellule.
L’intégration chromosomique est une forme de « génotoxicité », c’est-à-dire une toxicité qui provoque des dommages génétiques. En ce qui concerne la possibilité de tels effets, le rapport d’évaluation de l’EMA sur le vaccin ARNm de Pfizer note succinctement[4, p. 50] :
Aucune étude de génotoxicité n’a été fournie. Ceci est acceptable puisque les composants de la formulation du vaccin sont des lipides et de l’ARN qui ne devraient pas avoir de potentiel génotoxique.
Apparemment, les experts de l’EMA ont supposé que l’ARN en général n’affectera pas l’intégrité du génome de la cellule hôte. Ce point de vue est erroné, et la première preuve qui l’a démontré a récemment fêté son cinquantième anniversaire[27]. Cependant, la détection de quantités importantes d’ADN plasmidique dans les vaccins des deux fabricants rend désormais inutile cette argumentation. Même les scientifiques de l’EMA savent certainement que cet ADN peut être intégré dans le génome des cellules humaines hôtes. Aucune caractéristique de séquence spécifique n’est requise pour qu’une telle intégration se produise et, par conséquent, elle a été observée de la même manière avec l’ADN de virus de mammifères, les bactériophages et les plasmides[28]. Il convient de noter que ces insertions peuvent se produire à des endroits aléatoires du génome, mais les gènes activement exprimés par la cellule sont plus souvent affectés[29].
L’intégration chromosomique stable d’un plasmide bactérien dans l’ADN chromosomique de cellules de mammifères a été démontrée dès 1982[30]. Le plasmide en question partage de multiples caractéristiques avec ceux utilisés dans la production des vaccins ARNm de Moderna et Pfizer. L’introduction de gènes étrangers ou modifiés dans des cellules de mammifères à l’aide de cette technique et d’autres techniques similaires est devenue courante dans la recherche expérimentale et la biotechnologie. La méthodologie est appelée transfection, et les organismes modifiés de cette manière sont appelés transgènes. Il convient de noter qu’une intégration stable peut se produire avec de l’ADN plasmidique linéaire ou circulaire[31].
Dans ce contexte, nous devrions également tenir compte de l’étude publiée précédemment par Aldén et al[32], qui ont détecté des copies d’ADN du gène de la protéine spike dans une lignée de cellules hépatiques humaines après que ces cellules aient été exposées au vaccin ARNm de Pfizer. Partant de l’hypothèse que le vaccin contenait essentiellement de l’ARNm pur et non de l’ADN, ils ont considéré que cette observation prouvait que l’ARNm synthétique avait subi une transcription inverse dans ces cellules. Leur interprétation est plausible, car on sait qu’une telle transcription inverse se produit en principe et qu’elle a déjà été signalée dans des cellules de patients infectés par le virus SARS-CoV-2[33]. Cependant, à la lumière de la découverte de McKernan selon laquelle les flacons de vaccins Pfizer peuvent contenir des quantités substantielles d’ADN, il semble tout aussi possible que les observations d’Aldén et al. indiquent simplement l’absorption cellulaire de cet ADN. Quoi qu’il en soit, leurs conclusions indiquent la présence d’ADN codant pour la protéine Spike dans ces cellules, ce qui indique un risque d’insertion génomique.
4.3.1 Insertion génomique dans la thérapie génique utilisant des vecteurs rétroviraux
Dans le cadre d’une thérapie génique proprement dite, l’intégration chromosomique est souvent souhaitée, car elle permet de corriger durablement l’anomalie génétique en question. À cette fin, des vecteurs d’ADN spéciaux ont été mis au point, qui ont une propension beaucoup plus élevée à subir une telle intégration. Ces vecteurs sont dérivés des rétrovirus, dont toute la stratégie de survie repose sur l’intégration génomique. Il s’avère cependant que l’intégration, lorsqu’elle se produit au mauvais endroit dans le génome, induit souvent des maladies malignes, en particulier la leucémie[34]. Ce phénomène est en fait si courant qu’il a empêché l’adoption généralisée de la thérapie génique, même dans des maladies pour lesquelles toutes les autres options thérapeutiques sont également entachées de risques très graves. Un bon exemple est le déficit en adénosine désaminase, une maladie métabolique qui élimine les lymphocytes, provoquant ainsi un déficit immunitaire combiné sévère (SCID), une condition qui, sans traitement, est toujours fatale pendant l’enfance. Cette maladie est en principe une cible très appropriée pour la thérapie génique, mais une greffe de moelle osseuse provenant d’un donneur compatible et de la même famille reste l’option thérapeutique préférée en raison du risque important de tumeurs malignes induites par la thérapie génique[35].
4.3.2 Comment l’insertion génomique provoque-t-elle des tumeurs malignes ?
Notre génome contient de nombreux gènes qui peuvent donner lieu à un cancer si leur niveau d’expression – la vitesse à laquelle les molécules d’ARNm et de protéines sont synthétisées à partir de ces gènes – est trop faible ou trop élevé. Une molécule d’ADN étrangère peut s’insérer directement dans un tel gène et l’éliminer complètement, ou s’insérer à côté, et un promoteur puissant présent dans cet ADN étranger peut entraîner une surexpression du gène en question. En outre, il a été observé que les événements d’insertion peuvent également entraîner des modifications de la méthylation de l’ADN à l’échelle du génome, ce qui affectera les niveaux d’expression de nombreux gènes ; et certaines de ces modifications peuvent contribuer à l’induction d’une malignité. Il est important de noter que cet effet a été observé non seulement dans l’ADN viral, mais aussi dans les plasmides bactériens[36].
Lorsque des cellules sont isolées à partir d’un organe humain ou animal sain et cultivées, elles se divisent pendant un nombre limité de générations, puis meurent. En revanche, les cellules dérivées de tumeurs malignes et de leucémies peuvent se propager indéfiniment. Un changement similaire peut également se produire dans les cellules cultivées, qui deviennent alors immortalisées et perdent généralement certaines caractéristiques de leur tissu d’origine. Cette transformation peut être induite, par exemple, en infectant les cellules avec le virus SV40 susmentionné. De même, les cellules peuvent être transformées par transfection avec un plasmide dérivé du SV40 qui conserve les parties cruciales du génome viral, y compris le gène codant pour l’antigène T de grande taille. En revanche, si le grand antigène T est absent du plasmide, la transformation ne se produit généralement pas[30]. Cependant, quelques exceptions ont été signalées[37,38]. Ces cas doivent résulter de la perturbation ou de la régulation négative de gènes cellulaires impliqués dans le contrôle de la prolifération.
4.3.3 Intégration génomique dans les cellules germinales
Les ovocytes peuvent être transfectés in vivo à certains stades de maturation[39], de même que les cellules productrices de spermatozoïdes dans les testicules[40]. Dans ce dernier cas, la progéniture des animaux soumis à un tel traitement s’est révélée transgénique. On ne peut donc pas exclure que des personnes auxquelles on a injecté des vaccins à ARNm contenant également de l’ADN donnent ensuite naissance à des enfants transgéniques. L’insertion d’ADN dans les cellules germinales peut également interférer avec le développement intra-utérin précoce et provoquer ainsi des avortements spontanés ou des malformations.
4.3.4 Comment évaluer le risque d’insertion génomique ?
Il est certain que les plasmides bactériens ont une propension plus faible à s’insérer dans notre ADN chromosomique que les vecteurs de thérapie génique spécialement conçus pour une intégration efficace. Mais quelle est l’importance du risque dans le cas des plasmides contenus dans les vaccins à ARNm ? La réponse est simple : personne ne le sait. Ce n’est pas parce qu’il est inconnu en principe, mais parce que les études expérimentales appropriées sur les animaux, et ensuite sur les humains, n’ont pas été réalisées ; ou si elles l’ont été, les résultats ont été cachés au public, et apparemment aussi aux autorités de réglementation.
Comment ces risques seraient-ils évalués dans le cadre de procédures d’approbation correctement menées ? Les orientations actuelles de la FDA sur les essais et l’approbation des thérapies géniques[41] recommandent que, dans la phase d’essai clinique, les patients soient suivis pendant 15 années complètes après l’administration, avec des examens annuels au cours des cinq premières années. Cela s’applique aux vecteurs pour lesquels l’insertion chromosomique est prévue. Le document d’orientation établit une fausse dichotomie entre les vecteurs d’insertion et les vecteurs de non-insertion, mais la ligne de démarcation entre les deux reste floue. D’une part, le document d’orientation suggère que
Les produits de GT [thérapie génique] basés sur des vecteurs tels que les plasmides […] ne sont pas susceptibles de s’intégrer ou de se réactiver après une période de latence et présentent généralement un risque plus faible d’effets indésirables retardés,
mais, d’un autre côté, elle déclare que
des changements dans les méthodes utilisées pour introduire des vecteurs d’ADN plasmidique dans les cellules … entraînent des fréquences d’intégration plus élevées (Réf. 27).
La référence citée dans cette dernière citation est une étude de Wang et al[42], qui a identifié sans équivoque l’insertion d’ADN plasmidique in vivo après une injection intramusculaire, suivie d’une électroporation. Bien que l’électroporation ait augmenté l’absorption cellulaire de l’ADN injecté par rapport à l’injection d’ADN « nu » seul, elle était probablement beaucoup moins efficace à cet égard que les nanoparticules lipidiques contenues dans les vaccins ARNm. Par conséquent, il faut s’attendre à une certaine intégration chromosomique de l’ADN plasmidique contaminant in vivo.
4.4. effet pro-inflammatoire de l’ADN bactérien
Le système immunitaire inné humain réagit par l’inflammation à diverses macromolécules bactériennes, y compris l’ADN. Les grandes quantités d’ADN présentes dans les vaccins devraient être considérées comme contribuant à l’inflammation près du site d’injection, et potentiellement aussi ailleurs dans le corps.
5. Conclusion
La présence d’ADN plasmidique contaminant dans les vaccins ARNm de Pfizer et Moderna entraîne de graves risques pour la santé, en plus de ceux qui étaient déjà connus et compris. Ces risques comprennent l’expression prolongée de la protéine spike, qui peut conduire à une inflammation auto-immune correspondante et plus destructrice, et l’induction d’une maladie maligne à la suite de l’intégration chromosomique de l’ADN plasmidique. En outre, l’ampleur de la contamination prouve de manière irréfutable que les fabricants n’ont pas maîtrisé ou correctement mis en œuvre les processus de production prévus. Chacun de ces problèmes constituerait à lui seul une raison suffisante pour exiger le rappel immédiat de ces vaccins.
Remerciements
Nous remercions Kevin McKernan et Ulrike Kämmerer pour les corrections et les discussions.
Droits d’auteur
Ce texte est soumis aux termes de la licence Creative Commons Attribution 4.0 International License (CC BY 4.0). Cela signifie que vous êtes libre de copier et de réutiliser le contenu, à condition que les auteurs originaux soient cités. Si vous apportez des modifications au texte, vous devez l’indiquer explicitement. Pour plus de détails, consultez le site web de Creative Commons[43].
Références
- Andries, O. et al. (2015) N1-methylpseudouridine-incorporated mRNA outperforms pseudouridine-{}incorporated mRNA, providing enhanced protein expression and reducing immunogenicity in mammalian and mouse cell lines. J. Control.Release 217:337-344
- Anonyme, (2020) FDA briefing document : Pfizer-BioNTech COVID-19 vaccine.
- Anonyme, (2020) FDA briefing document : modern MRNA-1273.
- Anonyme, (2021) Rapport d’évaluation de l’EMA : Comirnaty.
- Anonyme, (2021) Rapport d’évaluation de l’EMA : COVID-19 Moderna Vaccine.
- McKernan, K. (2023) Deep sequencing of Moderna and Pfizer bivalent vaccines identifies contamination of expression vectors designed for plasmid amplification in bacteria.
- McKernan, K. (2023) Les vaccins bivalents de Pfizer et de Moderna contiennent 20 à 35 % de vecteurs d’expression et sont capables de transformer E.coli.
- McKernan, K. (2023) Contamination par l’ADN de 8 flacons de vaccins monovalents Pfizer à ARNm.
- Patel, H.K. et al. (2023) Characterization of BNT162b2 mRNA to Evaluate Risk of Off-Target Antigen Translation. J. Pharm.Sci. DOI:10.1016/j.xphs.2023.01.007
- Pardi, N. et al. (2018) Nucleoside-modified mRNA vaccines induce potent T follicular helper and germinal center B cells responses. J. Exp. Med. 215:1571-1588
- Bansal, S. et al. (2021) Cutting Edge : Circulating Exosomes with COVID Spike Protein Are Induced by BNT162b2 (Pfizer-BioNTech) Vaccination prior to Development of Antibodies : Un nouveau mécanisme d’activation immunitaire par les vaccins ARNm. J. Immunol. 207:2405-2410
- Castruita, J.A.S. et al. (2023) SARS-CoV-2 spike RNA vaccine sequences circulate in blood up to 28 days after COVID-19 vaccination. APMIS 131:128-132
- Fertig, T.E. et al. (2022) Vaccine mRNA can be detected in blood at 15 days post-vaccination. Biomedicines 10:1538
- Magen, E. et al. (2022) Clinical and Molecular Characterization of a Rare Case of BNT162b2 mRNA COVID-19 Vaccine-Associated Myositis (Caractérisation clinique et moléculaire d’un cas rare de myosite associée au vaccin BNT162b2 mRNA COVID-19). Vaccines 10
- Röltgen, K. et al. (2022) Immune imprinting, amplitude of variant recognition and germinal center response in human SARS-CoV-2 infection and vaccination. Cell DOI:10.1016/j.cell.2022.01.018
- Miao, C.H. et al. (2001) Long-term and therapeutic-level hepatic gene expression of human factor IX after naked plasmid transfer in vivo. Mol. Ther. 3:947-57
- Ye, X. et al. (2003) Complete and sustained phenotypic correction of hemophilia B in mice after hepatic gene transfer of a high expression human factor IX plasmid. J. Thromb. Haemost. 1:103-11
- Jager, L. et Ehrhardt, A. (2009) Persistence of high-throughput adenoviral vectors as monomeric replication-defective genomes in vitro and in murine liver. Hum.Gene Ther. 20:883-96
- Li, Y.Q. et al. (2000) [La fonction du promoteur T7 en tant qu’éléments cis-acting pour la polymérase II dans les cellules eucaryotes]. Yi Chuan Xue Bao 27:455-61
- Mörz, M. (2022) A Case Report : Encéphalite nécrosante et myocardite multifocale après la vaccination par l’ARNm BNT162b2 contre le Covid-19. Vaccines 10:2022060308
- Byrne, B.J. et al. (1983) Definition of the simian virus 40 early promoter region and demonstration of a host range bias in the enhancement effect of the simian virus 40 72-base-pair repeat. Proc.Natl.Acad.Sci.U. S. A. 80:721-5
- Rotondo, J.C. et al. (2019) Association entre le virus simien 40 et les tumeurs humaines. Front.Oncol. 9:670
- DeCaprio, J.A. et Garcea, R.L. (2013) A cornucopia of human polyomaviruses. Nat.Rev. Microbiol. 11:264-76
- Hussain, I. et al. (2020) Human BK and JC polyomavirus : molecular insights and prevalence in Asia. Virus Res. 278:197860
- La Bella, F. et Ozer, H.L. (1985) Differential replication of SV40 and polyoma DNAs in Chinese hamster ovary cells. Virus Res. 2:329-44
- Ehrhardt, A. et al. (2003) Episomal persistence of recombinant adenoviral vector genomes during the cell cycle in vivo. J. Virol. 77:7689-95
- Baltimore, D. (1970) RNA-dependent DNA polymerase in RNA tumor virus virions. Nature 226:1209-11
- Doerfler, W. (2016) Beware of genome manipulations : epigenetic destabilization via (foreign) DNA insertions. Epigenomics 8:587-91
- Doerfler, W. (1996) A new concept in (adenoviral) oncogenesis : foreign DNA integration and its consequences. Biochim.Biophys.Acta 1288:F79-99
- Southern, P.J. and Berg, P. (1982) Transformation of mammalian cells to antibiotic resistance with a bacterial gene under control of the SV40 early region promoter. J. Mol. Appl. Genet. 1:327-41
- Stuchbury, G. et Münch, G. (2010) Optimizing the generation of stable neuronal cell lines by pre-transfection restriction enzyme digestion of plasmid DNA. Cytotechnology 62:189-94
- Aldén, M. et al. (2022) Intracellular Reverse Transcription of Pfizer BioNTech COVID-19 mRNA Vaccine BNT162b2 In Vitro in Human Liver Cell Line. Curr.Emite Mol.Biol. 44:1115-1126
- Zhang, L. et al. (2020) SARS-CoV-2 RNA transcrit et intégré dans le génome humain. bioRxiv DOI:10.1101/2020.12.12.422516
- Staal, F.J.T. et al. (2008) Sola dosis facit venenum. La leucémie dans les essais de thérapie génique : une question de vecteurs, d’inserts et de dosage ? Leukemia 22:1849-1852
- Kohn, D.B. et Gaspar, H.B. (2017) How We Manage Adenosine Deaminase-Deficient Severe Combined Immune Deficiency (ADA SCID). J. Clin. Immunol. -n/a
- Doerfler, W. et al. (2018) Inheritable epigenetic response towards foreign DNA entry by mammalian host cells : a guardian of genomic stability. Epigenetics 13:1141-1153
- Sipehia, R. et Martucci, G. (1995) High-efficiency transformation of human endothelial cells by Apo E-mediated transfection with plasmid DNA. Biochem.Biophys.Commun.Res. 214:206-11
- Takahashi, M. et al. (2002) Transformation des cellules MC3T3-E1 après stress et transfection avec le plasmide pSV2neo. Anticancer Res. 22:585-98
- Laurema, A. et al. (2003) Transfection d’ovocytes et d’autres types de cellules ovariennes chez la lapine après injection directe dans les artères utérines d’adénovirus et de plasmides/liposomes. Gene Ther. 10:580-4
- Dhup, S. et Majumdar, S.S. (2008) Transgenesis through permanent gene integration in spermatogonial cell repopulation in vivo. Nat.Methods 5:601-3
- Anonyme, (2020) Long-term Follow-Up after administration of human gene therapy products : Guidance for Industry.
- Wang, Z. et al. (2004) Detection of integration of plasmid DNA into host genomic DNA following intramuscular injection and electroporation. Gene Therapy. 11:711-21
- Anonyme, (2023) Creative Commons Attribution 4.0 International License (CC BY 4.0).
Publié à l’origine dans Doctors for Covid Ethics
Suggérer une correction