COVID-19 mRNA-Impfstoffe enthalten übermäßige Mengen an bakterieller DNA: Beweise und Implikationen
Jüngste Studien von Kevin McKernan, einem führenden Experten für Sequenzierungsmethoden für DNA und RNA, haben ergeben, dass Chargen der modifizierten mRNA-Impfstoffe, die sowohl von Pfizer als auch von Moderna hergestellt werden, einen hohen Anteil an kontaminierender bakterieller DNA enthalten. Insgesamt macht die DNA bis zu 20-35 % der in den einzelnen Impfstoffchargen enthaltenen Nukleinsäuren aus. Diese alarmierend hohen Konzentrationen liegen weit über den Werten, die von normgebenden Organisationen wie der Europäischen Arzneimittelagentur (EMA) als sicher angesehen werden. Dieses Dokument fasst die Beweise für diese DNA-Kontamination zusammen und erörtert die möglichen Gesundheitsrisiken für die Empfänger der Impfstoffe.
1. Die Rolle der DNA bei der Herstellung von mRNA-Impfstoffen
1.1. Allgemeiner Hintergrund
Den meisten Lesern wird bekannt sein, dass
- die in den COVID-19-mRNA-Impfstoffen enthaltenen synthetischen RNAs für das SARS-CoV-2-Spike-Protein kodieren;
- in lebenden Säugetierzellen sind die Anweisungen für den Bau eines bestimmten Proteinmoleküls als Gen in der DNA im Zellkern gespeichert;
- um ein bestimmtes Eiweißmolekül zu bauen, schreibt die Zelle zunächst das Gen in RNA um und modifiziert die beiden Enden dieses Moleküls, um Boten-RNA (mRNA) zu bilden. Die mRNA wird dann vom Zellkern ins Zytoplasma transportiert, wo sie die Proteinfabriken der Zelle – die Ribosomen –dazu veranlasst, die Nukleotidsequenz der mRNA in die entsprechende Aminosäuresequenz zu übersetzen und das Protein zusammenzubauen.
1.2. Schritte bei der Herstellung von mRNA-Impfstoffen
Da das Spike-Protein ein großes Molekül ist, ist es auch die mRNA, die es kodiert. Die chemische Totalsynthese großer mRNA-Moleküle ist in großem Maßstab nicht praktikabel. Um das mRNA-Molekül, das für Spike kodiert, zu erhalten, wird daher der Prozess, mit dem Zellen ihre eigenen mRNAs produzieren, in vitro nachgeahmt. Dies umfasst die folgenden Schritte:
- Eine DNA-Kopie des Gens für das Spike-Protein wird in ein bakterielles Plasmid eingefügt. Dabei handelt es sich um ein ringförmiges, doppelsträngiges DNA-Molekül, das in einer Bakterienzelle unabhängig von der zelleigenen chromosomalen DNA existieren kann und das bei einer Zellteilung auch kopiert und an die beiden Tochterzellen weitergegeben werden kann.
- Das rekombinante (künstliche) Plasmid, das das Spike-Protein-Gen trägt, wird in eine Zelle der Bakterienart Escherichia coli(E. coli ) eingeführt. Da sich E. coli-Zellen sehr schnell teilen, kann diese eine Zelle innerhalb kurzer Zeit zu einer sehr großen Anzahl von Zellen heranwachsen. Es besteht zwar eine gewisse Wahrscheinlichkeit, dass das Plasmid bei aufeinanderfolgenden Zellteilungen aus einigen der Nachkommen verloren geht, aber wir können seine Erhaltung erzwingen, indem wir es mit einem selektiven Marker versehen, der dafür sorgt, dass nur die Zellen überleben, die das Plasmid behalten. Bei den Plasmiden, die sowohl von Pfizer als auch von Moderna verwendet werden, ist dieser Selektionsmarker ein Gen, das die Wirtszellen mit einer Resistenz gegen das Antibiotikum Kanamycin ausstattet. Um die Selektion durchzuführen, werden die Bakterien einfach in Gegenwart von Kanamycin gezüchtet.
- Nachdem eine ausreichende Anzahl von Bakterienzellen in einer Kanamycin-haltigen Nährbrühe herangewachsen ist, werden diese Zellen aufgebrochen und die Plasmid-DNA von den anderen Bestandteilen der Bakterienzellen gereinigt.
- Die ringförmigen Plasmidmoleküle werden mit Hilfe eines Restriktionsenzyms in eine lineare Form umgewandelt, das beide Stränge des DNA-Moleküls an einer spezifischen, einzigartigen Stelle spaltet, die sich stromabwärts des Spike-Protein-Gens befindet. Die linearisierten DNA-Moleküle können von den verbleibenden zirkulären Molekülen gereinigt werden, aber auf welche Weise und wie effizient dies bei der Herstellung der Impfstoffe von Pfizer und Moderna geschehen kann, ist nicht bekannt.
- Eine RNA-Polymerase wird in Gegenwart der erforderlichen Nukleosidbausteine und Cofaktoren verwendet, um das Spike-Protein-Gen von der DNA-Version auf dem linearisierten Plasmid in die mRNA-Version zu kopieren. Sowohl Pfizer als auch Moderna verwenden die T7-RNA-Polymerase, die von dem gleichnamigen Bakteriophagen abgeleitet ist. Dieses Enzym bindet an eine kognitive Promotorsequenz, die ebenfalls von T7 stammt und in das Plasmid stromaufwärts des Gens für das Spike-Protein eingebaut worden ist. In diesem Stadium wird das synthetische Nukleosid N-Methyl-Pseudouridin (mψU) anstelle des natürlichen Nukleosids Uridin in die künstliche RNA eingebaut. Wenn sie in Form eines Impfstoffs verabreicht wird, wirkt die auf diese Weise modifizierte RNA weniger stimulierend auf das angeborene Immunsystem als eine RNA, die das natürliche Uridin enthält. Außerdem wird sie effizienter in Proteine übersetzt und ist unter bestimmten Bedingungen resistenter gegen den Abbau[1]. Sowohl die mRNA-Impfstoffe von Pfizer als auch die von Moderna enthalten mψU anstelle von Uridin.
- Die beiden Enden jedes RNA-Moleküls werden enzymatisch an bestimmte Einheiten gekoppelt, die sich auch in natürlichen Säugetier-mRNAs an diesen Stellen befinden und die biologische Aktivität und Stabilität in vivo erhöhen.
Auf diese Weise entsteht eine funktionsfähige mRNA, die die Ribosomen der Zellen anweisen kann, das Spike-Protein zu produzieren. In diesem Stadium ist das Produkt jedoch noch nicht rein – die bakteriell gewonnene Template-DNA ist noch vorhanden. Letztere sollte nicht in das endgültige Arzneimittel aufgenommen werden, da sie ein Gesundheitsrisiko für die Empfänger darstellt (siehe Abschnitt 4). Um diese DNA loszuwerden, wird ein weiteres Enzym namens DNase hinzugefügt. Dieses soll die DNA in kleinere Fragmente aufspalten, die dann durch Filtration und andere Reinigungstechniken von den viel größeren RNA-Molekülen getrennt werden können. Im letzten Schritt wird die mRNA mit einer Lipidmischung kombiniert, um sie in Lipid-Nanopartikel (LNPs) zu verpacken, die menschliche Zellen dazu veranlassen, das mRNA-Molekül aufzunehmen und das Spike-Protein herzustellen.
2. Was wussten wir bisher über das Problem der DNA-Kontamination?
Kurz gesagt, sehr wenig. In den Bewertungsberichten der FDA zu beiden Impfstoffen[2,3] wird das Problem mit keinem Wort erwähnt. Im Bewertungsbericht der Europäischen Arzneimittelagentur (EMA) über den Impfstoff von Pfizer heißt es: „Die Robustheit des DNase-Verdauungsschritts wird nicht als umfassend nachgewiesen angesehen“[4, S. 17]. Eine ähnliche Formulierung findet sich im EMA-Bericht über den Moderna-Impfstoff[5, S. 19f]. Allein auf der Grundlage dieser spärlichen Informationen ist es jedoch unmöglich zu sagen, ob das Problem als schwerwiegend angesehen wurde und welche Abhilfemaßnahmen von der Aufsichtsbehörde gegebenenfalls gefordert wurden.
3. Unabhängige Beweise für die DNA-Kontamination von mRNA-Produkten
Mit Stand vom 3. April 2023 hat Kevin McKernan seine jüngsten Erkenntnisse in drei Artikeln auf seiner Substack-Seite beschrieben[6-8]. Die in den ersten beiden Berichten beschriebenen Experimente wurden an Proben von neu eingeführten „bivalenten“ Impfstoffen von Pfizer und Moderna durchgeführt. Diese Präparate ähneln in ihrer chemischen Zusammensetzung den bisherigen „monovalenten“, d.h. sie sollten hochreine mRNA enthalten, die mit einer Mischung aus Lipidmolekülen (fettähnlich) zu mRNA/Lipid-Nanopartikeln komplexiert ist. Der einzige Unterschied zwischen den beiden Varianten ist, dass die bivalenten Impfstoffe eine Mischung aus zwei mRNAs enthalten, die für zwei antigene Varianten des Spike-Proteins kodieren. Dies hat keinen Einfluss auf das technische Problem der DNA-Kontamination als solches. Wir weisen jedoch darauf hin, dass das Ausmaß der DNA-Kontamination zwischen den Produktionschargen variieren kann und dass bisher nur eine kleine Anzahl von Chargen in dieser Hinsicht charakterisiert wurde.
3.1. Der erste Bericht von McKernan
In einer ersten Studie[6] charakterisierte McKernan sowohl die in den mRNA-Impfstoffen enthaltene RNA als auch die DNA.
3.1.1. Extraktion und direkte Charakterisierung der Nukleinsäuren aus den Impfstoffen
Der erste Schritt bestand darin, die Lipide abzulösen, um die reinen Nukleinsäuren zu erhalten. Die von ihm verwendete lösungsmittelbasierte Methode unterscheidet nicht zwischen DNA und RNA – wenn beide vorhanden sind, werden auch beide gewonnen. Die extrahierten Nukleinsäuren wurden nach ihrer Größe getrennt. Dabei wurden nicht nur die erwarteten regulären Spike-mRNA-Arten in voller Länge gefunden, sondern auch kleinere Fragmente, die zuvor sowohl von den Regulierungsbehörden als auch in einer von einem der Hersteller veröffentlichten Arbeit festgestellt worden waren[9]. Überraschenderweise wurden auch RNA-Spezies gefunden, die größer sind als die mRNA in voller Länge. Diese Spezies bleiben uncharakterisiert.
3.1.2. Amplifikation der extrahierten Nukleinsäuren
Als vorbereitender Schritt für die Bestimmung der genauen Nukleotidsequenzen der extrahierten Nukleinsäuren wurden diese durch PCR-Methoden amplifiziert. Im Falle der RNA ging der PCR eine reverse Transkription in DNA unter Verwendung eines speziellen Enzyms (reverse Transkriptase) voraus. Da in dieser Studie in erster Linie die RNA und nicht die DNA untersucht werden sollte, wurde dieser Vervielfältigungsschritt durch Zugabe von Actinomycin D, das unter den gegebenen Versuchsbedingungen die DNA-Synthese selektiv hemmt, auf die DNA ausgerichtet. Dementsprechend wurden relativ geringe Mengen an DNA in der amplifizierten Probe gewonnen. Im Falle des Pfizer-Impfstoffs überstieg die festgestellte DNA-Menge jedoch bereits den von der EMA willkürlich festgelegten Grenzwert für den maximal zulässigen Anteil von DNA pro RNA.
3.1.3. Ergebnisse der DNA-Sequenzierung
Sowohl für die Produkte von Pfizer als auch für die von Moderna wurden DNA-Sequenzen von vollständigen DNA-Plasmiden erhalten, wobei im Falle der Moderna-Plasmide eine gewisse Unklarheit bestehen blieb. Die Merkmale der Plasmidsequenzen werden daher im Zusammenhang mit der zweiten Studie von McKernan erörtert, bei der mehr und reinere DNA für die Sequenzierung verwendet wurde und die daher zuverlässigere Ergebnisse lieferte.
3.2. Der zweite Bericht von McKernan
Die zweite Studie[7] konzentrierte sich auf die Quantifizierung und Charakterisierung der DNA-Kontamination, die in der ersten Studie qualitativ festgestellt worden war.
3.2.1. Die in den mRNA-Impfstoffen enthaltene Plasmid-DNA ist in der Lage, sich in Bakterienzellen zu vermehren
Im ersten Versuch wurde festgestellt, ob die Plasmid-DNA, deren Vorhandensein aus den vorangegangenen Sequenzierungsergebnissen abgeleitet wurde, tatsächlich biologisch funktionsfähig ist, d. h., ob sie in Bakterienzellen eingeführt werden und dort verbleiben kann. Zu diesem Zweck wurden erneut Nukleinsäuren aus den Impfstoffproben extrahiert. Diese Nukleinsäuren wurden mit einer Suspension von E. coli-Zellen gemischt, die für die DNA-Aufnahme kompetent gemacht worden waren.
Nachdem diese Zellen zur DNA-Aufnahme veranlasst worden waren und sich erholt hatten, wurden sie auf Petrischalen ausgebreitet, die mit verfestigtem Kanamycin-haltigem Wachstumsmedium gefüllt waren. Wie bereits erwähnt, tötet Kanamycin alle E. coli-Zellen ab, die kein Resistenzgen dagegen enthalten. Daher bestätigte das beobachtete Wachstum von Bakterienkolonien auf diesen Petrischalen, dass einige Zellen tatsächlich eine Resistenz gegen Kanamycin erworben hatten, indem sie die Plasmide aufnahmen und vermehrten. Dies wurde sowohl bei den Impfstoffproben von Pfizer als auch von Moderna beobachtet.
In diesem Zusammenhang ist anzumerken, dass nur zirkuläre Plasmidmoleküle, nicht aber linearisierte, effizient in Bakterienzellen eingebracht werden können. Der Erfolg dieses Experiments lässt daher vermuten, dass einige der Plasmidmoleküle dem Linearisierungsschritt (Schritt 4 in Abschnitt 1.2) entgangen sind und den gesamten Produktionsprozess in der zirkulären Form, die in Bakterienzellen existiert, durchlaufen haben. Da die Zahl der in diesem Experiment beobachteten Bakterienkolonien jedoch nicht hoch war, ist es wahrscheinlich, dass der größte Teil der DNA tatsächlich linearisiert wurde. Da die biologischen Gefahren von Fremd-DNA in unserem eigenen Körper je nachdem, ob es sich um lineare oder zirkuläre DNA handelt, unterschiedlich sein können, ist das wahrscheinliche Vorhandensein beider Formen in den Impfstoffen erwähnenswert. Die genauen Anteile von zirkulärer und linearer DNA in den Mischungen müssen noch ermittelt werden.
3.2.2. Die Häufigkeit der kontaminierenden DNA
Das zweite wichtige Ergebnis dieser Studie ist die Quantifizierung der in den Impfstoffproben enthaltenen DNA und mRNA mittels PCR. Wie Sie vielleicht wissen, wird bei einer PCR-Reaktion ein ausgewählter Abschnitt einer Nukleinsäuresequenz durch enzymatische Synthese in mehreren aufeinanderfolgenden Reaktionszyklen vervielfältigt. Aus der Anzahl der Zyklen (oder Verdopplungen), die erforderlich sind, um eine bestimmte Schwellenkonzentration zu erreichen, lässt sich berechnen, wie viele Kopien der Zielsequenz zu Beginn vorhanden waren.
In diesen Experimenten wurde als Versuchsformat die Multiplex-PCR gewählt, d. h. zwei Zielsequenzen wurden in einer einzigen Reaktionsmischung amplifiziert. Eine dieser Zielsequenzen befand sich innerhalb des Spike-Protein-Gens und sollte daher sowohl auf den Plasmid-DNA-Molekülen als auch auf den von ihnen transkribierten Spike-mRNA-Molekülen vorhanden sein. Um die mRNA-Moleküle in diese Amplifikation einzubeziehen, wurde der PCR wiederum eine reverse Transkription vorgeschaltet.
Die andere Zielsequenz befand sich innerhalb des Kanamycin-Resistenzgens, das nur auf der Plasmid-DNA vorhanden sein sollte. Durch den Vergleich der Anzahl der Zyklen, die für jedes der beiden Ziele erforderlich sind, um den Schwellenwert zu überschreiten, wurde festgestellt, dass bis zu 35 % der gesamten in den Impfstoffen enthaltenen Nukleinsäure tatsächlich DNA ist. Zum Vergleich: Die EMA hat festgelegt, dass der Anteil der DNA an den gesamten Nukleinsäuren nicht mehr als 0,033 % betragen darf.
3.2.3. Bestimmung der DNA-Sequenzen der Plasmide
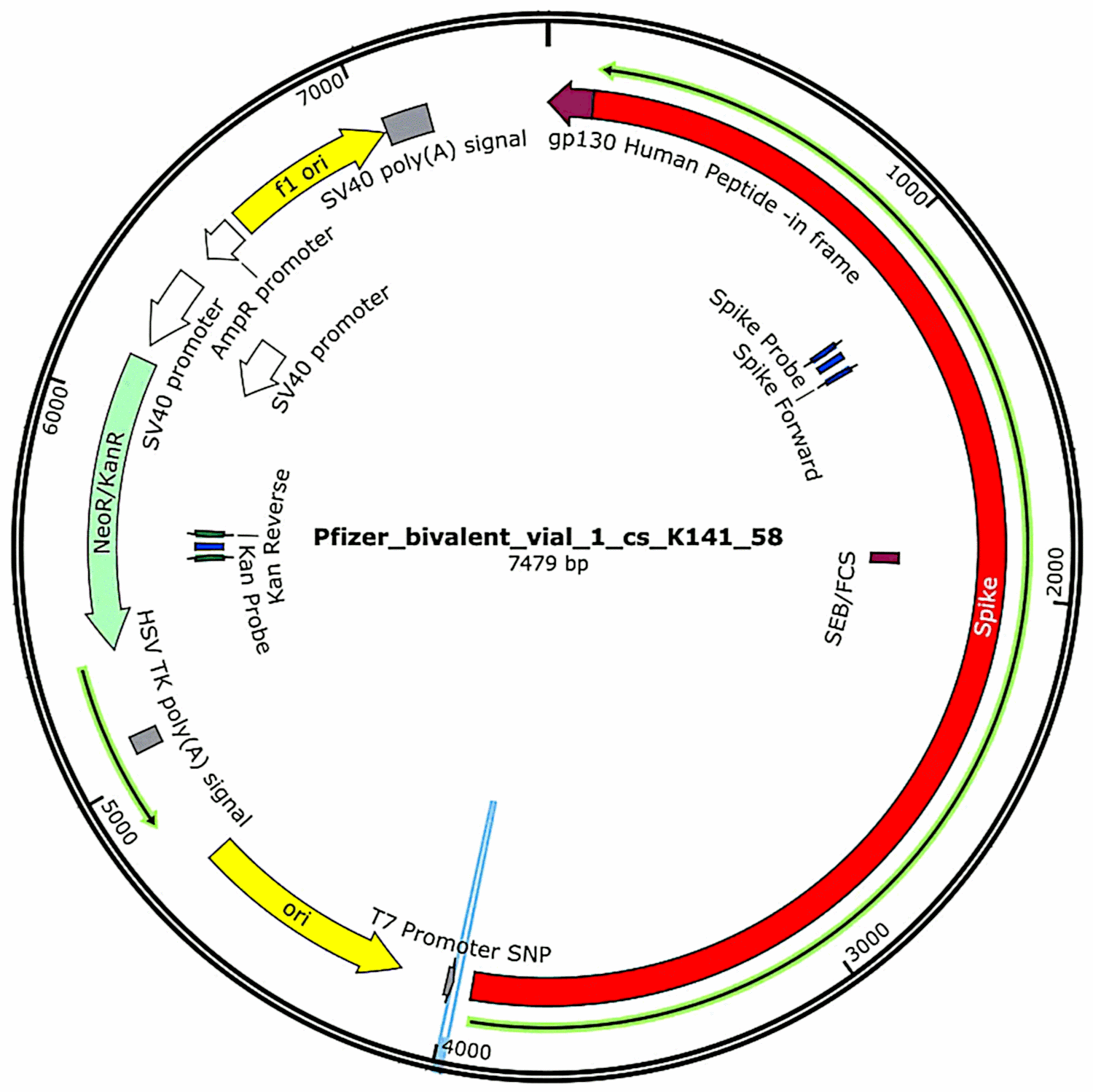
Die Plasmide, die ursprünglich in den Impfstoffen enthalten waren und dann in Bakterienzellen eingebracht wurden (siehe Abschnitt 3.2.1), wurden aus diesen Bakterienkulturen wieder isoliert und ihre vollständigen DNA-Sequenzen wurden bestimmt. Diese Sequenzen wurden in der ersten Studie von McKernan[6] vollständig angegeben, aber er gab an, dass er noch an der Bestätigung und Verfeinerung der Sequenzierungsdaten arbeitet. Die funktionellen Merkmale der in den Impfstoffproben von Pfizer gefundenen Plasmid-DNA sind in Abbildung 1 dargestellt. Sie werden im Zusammenhang mit der Risikobewertung erörtert.
3.3. Der dritte Bericht von McKernan
In seinem bisher letzten Bericht untersuchte McKernan acht Fläschchen einer früheren Charge des Pfizer-Impfstoffs mit der oben beschriebenen quantitativen PCR-Methode. Der DNA-Gehalt war in diesem Fall deutlich niedriger als bei den bivalenten Impfstoffproben, überschritt aber immer noch den EMA-Grenzwert um das 18-70fache[8].
4. Risikobewertung
Es ist davon auszugehen, dass die in den mRNA-Impfstoffen enthaltene rekombinante DNA in die Zellen unseres Körpers eingeschleust werden kann und dass dies, wie bei der mRNA selbst, durch die Lipid-Nanopartikel begünstigt wird. Dies birgt verschiedene Gesundheitsrisiken.
4.1. Längere Dauer der Expression des Spike-Proteins
Ein Hauptargument, das regelmäßig angeführt wird, um den Eindruck zu erwecken, dass mRNA-Impfstoffe sicher sind, ist, dass mRNA in vivo kurzlebig ist und dass die Expression des kodierten Antigens daher ebenfalls von kurzer Dauer sein wird. So heißt es beispielsweise im EMA-Bewertungsbericht über den Impfstoff von Pfizer in Bezug auf Tierversuche mit einem Modellimpfstoff, die anstelle angemessener Studien mit dem eigentlichen COVID-19-Impfstoff akzeptiert wurden[4, S. 46]:
Wie bei einem mRNA-Produkt zu erwarten, war die Luziferase-Expression vorübergehend … Das Signal nahm in den ersten 72 Stunden langsam ab, und nach 6 und 9 Tagen waren die Signale weiter abgeschwächt, bis sie etwa das 18- bzw. 7-fache der Signale von Tieren erreichten, denen eine Pufferkontrolle injiziert worden war.
Diese Ergebnisse scheinen mit zwei In-vitro-Studien übereinzustimmen, in denen die Dauer der Proteinexpression zwischen Boten-RNA-Spezies verglichen wurde, die in ihrer Sequenz identisch waren, aber Uridin bzw. mψU enthielten; wie bereits erwähnt, ist letzteres auch in den mRNA-Impfstoffen von Pfizer und Moderna enthalten. In beiden Studien[1,10] induzierten die mψU-modifizierten RNA-Spezies eine signifikant höhere Proteinexpression, dennoch nahm diese erhöhte Expression mit einer ähnlichen Halbwertszeit ab wie die der unmodifizierten RNA. Keine der Halbwertszeiten, die sich aus den Daten der beiden Studien ableiten lassen, überschreitet 4,5 Tage.
Aus mehreren Studien an geimpften Personen geht jedoch hervor, dass sowohl das Spike-Protein selbst als auch die dafür kodierenden Nukleinsäuren noch Wochen und sogar Monate nach der Injektion im Blutkreislauf und in verschiedenen Organen nachgewiesen werden können[11-15]. Diese Diskrepanz zwischen In-vitro- und In-vivo-Studien war bisher schwer zu verstehen. Die von McKernan festgestellten hohen Restmengen an Plasmid-DNA in den Impfstoffen legen nun eine plausible Erklärung nahe.
Damit die bakterielle Plasmid-DNA eine verlängerte Expression des Spike-Proteins unterstützen kann, müssen zwei Bedingungen erfüllt sein:
- die Plasmid-DNA muss in unseren Körperzellen verbleiben, und
- das Spike-Protein-Gen auf diesem Plasmid muss von unserer eigenen zellulären RNA-Polymerase II in mRNA umgeschrieben werden.
Wir haben zwar noch keine direkten experimentellen Daten zu den Spike-Expressionsplasmiden von Pfizer und Moderna, aber Präzedenzfälle deuten darauf hin, dass beide Anforderungen erfüllt sind. Es wurde festgestellt, dass rekombinante Plasmide, die den Gerinnungsfaktor IX exprimieren, in den Leberzellen von Versuchstieren bis zu 1,5 Jahre lang in stabilen Konzentrationen persistieren[16,17], was der gesamten Dauer des Versuchs entspricht. Man könnte einwenden, dass die in diesen Studien verwendeten Plasmide zirkulär waren, während der größte Teil der in den mRNA-Impfstoffen enthaltenen Plasmid-DNA wahrscheinlich in linearer Form vorliegt (siehe Abschnitt 1.2). Dazu ist anzumerken, dass erstens wahrscheinlich ein Teil der zirkulären Plasmid-DNA verbleibt (siehe Abschnitt 3.2.1) und zweitens rekombinante virale DNA in Tieren nachweislich ebenso lange in linearer Form erhalten bleibt[18], was darauf schließen lässt, dass dies auch bei Plasmid-DNA der Fall sein kann.
In den zitierten Studien[16,17] stand das Gen, das für das interessierende Protein (Faktor IX) kodiert, unter der Kontrolle eines Säugetierpromotors, und das Faktor-IX-Protein wurde tatsächlich durchgehend in stabilen Mengen exprimiert. Im Gegensatz dazu steht das Spike-Protein-Gen in den Expressionsplasmiden von Pfizer und Moderna unter der Kontrolle eines T7-Bakteriophagen-Promotors. Wir können nicht von vornherein davon ausgehen, dass dieser Promotor in Abwesenheit seiner kognitiven T7-RNA-Polymerase funktionieren wird. Es wurde jedoch experimentell bestätigt, dass der T7-Promotor auch die zelluläre RNA-Polymerase II bindet und die Proteinexpression in Säugetierzellen bewirkt[19].
Zusammenfassend lässt sich sagen, dass die Möglichkeit, dass die beobachtete lang anhaltende Expression des Spike-Proteins durch die in den mRNA-Impfstoffen enthaltene Plasmid-DNA verursacht wird, ernst genommen werden muss. Die in Biopsien und Autopsien nachgewiesene lang anhaltende Persistenz von Spike-Protein-mRNA und ihre Expression nach der Impfung wurde eindeutig mit schwerwiegenden Schäden in Verbindung gebracht[14,20], die höchstwahrscheinlich durch Immunangriffe auf die Zellen, die dieses fremde Antigen exprimieren, vermittelt werden. Das Fehlen entsprechender experimenteller Studien in der Phase der präklinischen Versuche in Verbindung mit dem Ausmaß dieser Kontamination stellt ein völlig inakzeptables Sicherheitsrisiko dar.
4.2. Risiken im Zusammenhang mit von SV40 abgeleiteten regulatorischen DNA-Sequenzen
Ein Merkmal, das McKernan auf den Expressionsplasmiden von Pfizer, nicht aber auf denen von Moderna identifiziert hat[6], ist ein vom SV40-Virus abgeleiteter Promotor, der zur Polyoma-Familie gehört (siehe Abschnitt 4.2). Dieser Promotor befindet sich stromaufwärts des Kanamycin-Resistenzgens; und da er in Säugetierzellen aktiv ist, wird das von diesem Resistenzgen kodierte Protein in jeder Zelle, die diese DNA enthält, exprimiert. Wie das Spike-Protein ist auch dieses Protein ein fremdes Antigen und kann daher ebenfalls einen Immunangriff auf die Zellen auslösen, die es exprimieren.
Der SV40-Promotor enthält auch einen internen Replikationsursprung, der dazu führen kann, dass Kopien des Plasmids in Säugetierzellen gebildet werden[21]. Dazu muss das virale große T-Antigen vorhanden sein, ein Protein, das diesen Ursprung direkt erkennt und dann die Replikation des DNA-Moleküls einleitet. Dieses Protein wird weder vom Plasmid kodiert, noch ist es normalerweise in unseren Körperzellen vorhanden, aber es könnte entweder vom SV40-Virus selbst oder von einem verwandten Polyoma-Virus geliefert werden. Eine Minderheit der menschlichen Bevölkerung ist latent mit SV40 infiziert, und eine solche latente Infektion wird mit einigen bösartigen und nicht bösartigen Krankheiten in Verbindung gebracht[22]. Sollte eine Kopie des Pfizer-Plasmids in eine Zelle aufgenommen werden, die SV40 beherbergt, könnten in der Tat weitere Kopien des Plasmids gebildet werden.
Zwei verwandte Polyoma-Viren, die in der menschlichen Bevölkerung viel weiter verbreitet sind, sind das BK- und das JC-Virus[23,24]. Das große T-Antigen des JC-Virus ist in Verbindung mit dem SV40-Ursprung offenbar weniger wirksam als das SV40-eigene Protein[25], aber die Replikation des Plasmids von Pfizer in Zellen, die latent mit JC- oder BK-Viren infiziert sind, kann dennoch nicht ausgeschlossen werden. Zusätzliche Kopien des auf diese Weise erzeugten Plasmids würden alle anderen in diesem Abschnitt diskutierten Risiken verstärken, mit der möglichen Ausnahme einer unspezifischen Entzündung (siehe Abschnitt 4.4).
4.3. Genomische Insertion der Plasmid-DNA
Bei allen bisher erörterten Szenarien handelt es sich um eine unabhängige episomale Persistenz der Plasmid-DNA, die in der Nähe der Chromosomen (innerhalb des Zellkerns) vorhanden ist, aber nicht zu einem integralen Bestandteil der Chromosomen geworden ist. Solche unabhängigen, nicht replizierenden Plasmidmoleküle gehen bei der Zellteilung tendenziell verloren[26]. Wie wir noch sehen werden, kann ein Plasmidmolekül jedoch in einigen Fällen tatsächlich in eines der Chromosomen seiner Wirtszelle integriert werden, und es wird dann an alle Nachkommen dieser Zelle vererbt.
Die chromosomale Integration ist eine Form der „Genotoxizität“, d. h. der Toxizität, die genetische Schäden verursacht. In Bezug auf die Möglichkeit solcher Effekte stellt der EMA-Bewertungsbericht zum mRNA-Impfstoff von Pfizer lapidar fest[4, S. 50]:
Es wurden keine Studien zur Genotoxizität vorgelegt. Dies ist akzeptabel, da es sich bei den Bestandteilen der Impfstoffformulierung um Lipide und RNA handelt, bei denen kein genotoxisches Potenzial zu erwarten ist.
Offenbar gingen die Experten der EMA davon aus, dass RNA im Allgemeinen die Integrität des Wirtszellgenoms nicht beeinträchtigt. Diese Ansicht ist falsch, und der erste Nachweis, der dies beweist, hat kürzlich sein fünfzigjähriges Jubiläum gefeiert[27]. Der Nachweis großer Mengen von Plasmid-DNA in den Impfstoffen beider Hersteller macht diese Behauptung nun jedoch überflüssig. Sicherlich ist auch den Wissenschaftlern der EMA bewusst, dass diese DNA in das Genom menschlicher Wirtszellen integriert werden kann. Für eine solche Integration sind keine spezifischen Sequenzmerkmale erforderlich, und dementsprechend ist sie auch bei der DNA von Säugetierviren, Bakteriophagen und Plasmiden beobachtet worden[28]. Es ist erwähnenswert, dass solche Einfügungen an beliebigen Stellen des Genoms auftreten können, aber Gene, die von der Zelle aktiv exprimiert werden, sind häufiger betroffen[29].
Die stabile chromosomale Integration eines bakteriellen Plasmids in die chromosomale DNA von Säugetierzellen wurde bereits 1982 nachgewiesen[30]. Das betreffende Plasmid weist mehrere Gemeinsamkeiten mit den Plasmiden auf, die bei der Herstellung der mRNA-Impfstoffe von Moderna und Pfizer verwendet werden. Das Einschleusen von fremden oder veränderten Genen in Säugetierzellen mit Hilfe dieser und ähnlicher Techniken ist in der experimentellen Forschung und in der Biotechnologie inzwischen gang und gäbe. Die Methode wird als Transfektion bezeichnet, und die auf diese Weise veränderten Organismen als transgene. Wir stellen fest, dass eine stabile Integration sowohl mit linearer als auch mit zirkulärer Plasmid-DNA erfolgen kann[31].
In diesem Zusammenhang ist auch die zuvor veröffentlichte Studie von Aldén et al.[32] zu berücksichtigen, die DNA-Kopien des Spike-Protein-Gens in einer menschlichen Leberzelllinie nachwiesen, nachdem diese Zellen dem mRNA-Impfstoff von Pfizer ausgesetzt worden waren. Ausgehend von der Annahme, dass der Impfstoff im Wesentlichen reine mRNA, aber keine DNA enthielt, werteten sie diese Beobachtung als Beweis dafür, dass die synthetische mRNA in diesen Zellen eine reverse Transkription durchlaufen hatte. Ihre Interpretation ist plausibel, da eine solche reverse Transkription prinzipiell bekannt ist und bereits in Zellen von Patienten, die mit dem SARS-CoV-2-Virus infiziert sind, beobachtet wurde[33]. In Anbetracht der Entdeckung von McKernan, dass Impfstoffampullen von Pfizer erhebliche Mengen an DNA enthalten können, scheint es jedoch ebenso möglich, dass die Beobachtungen von Aldén et al. einfach auf die zelluläre Aufnahme dieser DNA hinweisen. In jedem Fall deuten ihre Ergebnisse auf das Vorhandensein von Spike-kodierender DNA in diesen Zellen hin, was ein Risiko der genomischen Insertion bedeutet.
4.3.1. Genomische Insertion bei der Gentherapie mit retroviralen Vektoren
Bei der Gentherapie wird häufig eine chromosomale Integration angestrebt, um den betreffenden Gendefekt dauerhaft zu korrigieren. Zu diesem Zweck wurden spezielle DNA-Vektoren entwickelt, die eine stark erhöhte Neigung zu einer solchen Integration aufweisen. Diese Vektoren sind von Retroviren abgeleitet, deren gesamte Überlebensstrategie auf der genomischen Integration beruht. Es hat sich jedoch herausgestellt, dass die Integration, wenn sie an der falschen Stelle im Genom stattfindet, häufig bösartige Krankheiten, insbesondere Leukämie, auslöst[34]. Diese Tatsache ist so häufig, dass sie die breite Anwendung der Gentherapie verhindert hat, selbst bei Krankheiten, bei denen alle anderen therapeutischen Optionen ebenfalls mit sehr großen Risiken behaftet sind. Ein gutes Beispiel dafür ist der Adenosin-Deaminase-Mangel, eine Stoffwechselkrankheit, die die Lymphozyten auslöscht und damit eine schwere kombinierte Immunschwäche (SCID) verursacht, die ohne Behandlung im Säuglingsalter immer tödlich verläuft. Diese Krankheit ist prinzipiell ein sehr geeignetes Ziel für die Gentherapie, dennoch bleibt eine Knochenmarktransplantation von einem passenden und verwandten Spender die bevorzugte therapeutische Option aufgrund des großen Risikos von Gentherapie-induzierten Malignitäten[35].
4.3.2. Wie führt die genomische Insertion zu bösartigen Erkrankungen?
Unser Genom enthält eine Vielzahl von Genen, die Krebs auslösen können, wenn ihr Expressionsniveau – die Rate, mit der mRNA und Proteinmoleküle aus ihnen synthetisiert werden – entweder zu niedrig oder zu hoch ist. Ein fremdes DNA-Molekül kann sich möglicherweise direkt in ein solches Gen einfügen und es ganz ausschalten, oder es kann sich daneben einfügen, und ein starker Promotor auf dieser fremden DNA kann eine übermäßige Expression des fraglichen Gens verursachen. Darüber hinaus wurde beobachtet, dass Insertionsereignisse auch genomweite Veränderungen der DNA-Methylierung bewirken können, die sich auf die Expressionswerte vieler Gene auswirken; einige dieser Veränderungen können zur Auslösung von Malignität beitragen. Wichtig ist, dass dieser Effekt nicht nur bei viraler DNA, sondern auch bei bakteriellen Plasmiden beobachtet wurde[36].
Wenn Zellen aus einem gesunden menschlichen oder tierischen Organ isoliert und in einer Zellkultur gezüchtet werden, teilen sie sich für eine begrenzte Anzahl von Generationen und sterben dann ab. Im Gegensatz dazu können Zellen aus bösartigen Tumoren und Leukämien unbegrenzt vermehrt werden. Eine ähnliche Veränderung kann auch bei kultivierten Zellen eintreten, die dadurch unsterblich werden und typischerweise auch einige Merkmale verlieren, die für ihr Ursprungsgewebe charakteristisch sind. Diese Umwandlung kann z. B. durch Infektion der Zellen mit dem bereits erwähnten SV40-Virus ausgelöst werden. Ebenso können die Zellen durch Transfektion mit einem von SV40 abgeleiteten Plasmid transformiert werden, das die entscheidenden Teile des viralen Genoms, einschließlich des Gens, das für das große T-Antigen kodiert, beibehält. Fehlt hingegen das große T-Antigen auf dem Plasmid, kommt es in der Regel nicht zur Transformation[30]. Es wurden jedoch auch einige Ausnahmen berichtet[37,38]. Diese Fälle müssen durch die Störung oder Dysregulation von zellulären Genen entstanden sein, die an der Kontrolle der Proliferation beteiligt sind.
4.3.3. Genomische Integration in Keimbahnzellen
Eizellen können in bestimmten Reifungsstadien in vivo transfiziert werden[39], ebenso wie spermienproduzierende Zellen in den Hoden[40]. In letzterem Fall erwiesen sich die Nachkommen der so behandelten Tiere als transgen. Es ist daher nicht auszuschließen, dass Personen, die mit mRNA-Impfstoffen geimpft werden, die auch DNA enthalten, später transgene Kinder zur Welt bringen. Der DNA-Eintrag in Keimbahnzellen könnte auch die frühe intrauterine Entwicklung stören und dadurch Fehlgeburten oder Missbildungen hervorrufen.
4.3.4. Wie ist das Risiko einer genomischen Insertion zu bewerten?
Es ist sicherlich richtig, dass bakterielle Plasmide eine geringere Neigung haben, sich in unsere chromosomale DNA einzufügen, als Gentherapievektoren, die speziell für eine effiziente Integration entwickelt wurden. Aber wie groß ist das Risiko im Falle der in den mRNA-Impfstoffen enthaltenen Plasmide genau? Die einfache Antwort lautet: Niemand weiß es. Das liegt nicht daran, dass man es prinzipiell nicht wissen kann, sondern daran, dass die entsprechenden experimentellen Studien an Tieren und später am Menschen nicht durchgeführt wurden; oder wenn doch, wurden die Ergebnisse der Öffentlichkeit und offenbar auch den Regulierungsbehörden vorenthalten.
Wie würden solche Risiken in einem ordnungsgemäß durchgeführten Zulassungsverfahren bewertet werden? In den aktuellen FDA-Leitlinien für die Prüfung und Zulassung von Gentherapien[41] wird empfohlen, dass die Patienten in der Phase der klinischen Prüfung 15 Jahre lang nach der Verabreichung überwacht werden, wobei in den ersten fünf Jahren jährliche Untersuchungen vorgesehen sind. Dies gilt für Vektoren, mit denen eine chromosomale Insertion beabsichtigt ist. In dem Leitfaden wird eine falsche Dichotomie zwischen Insertionsvektoren und Nicht-Insertionsvektoren konstruiert, aber die Trennlinie zwischen beiden bleibt unscharf. Einerseits legt der Leitfaden nahe, dass
GT [Gentherapie]-Produkte, die auf Vektoren wie Plasmiden basieren … nicht zur Integration oder Reaktivierung nach einer Latenzzeit neigen, weisen im Allgemeinen ein geringeres Risiko für verzögerte unerwünschte Ereignisse auf,
andererseits heißt es aber auch, dass
änderungen bei den Methoden zur Einführung von Plasmid-DNA-Vektoren in Zellen … zu einer höheren Integrationshäufigkeit führen (Ref. 27).
Bei der im letztgenannten Zitat zitierten Referenz handelt es sich um eine Studie von Wang et al.[42], die eine DNA-Insertion von Plasmid-DNA in vivo nach intramuskulärer Injektion, gefolgt von Elektroporation, eindeutig nachweisen konnten. Die Elektroporation erhöhte zwar die zelluläre Aufnahme der injizierten DNA im Vergleich zur Injektion von „nackter“ DNA allein, war aber in dieser Hinsicht wahrscheinlich viel weniger wirksam als die in den mRNA-Impfstoffen enthaltenen Lipid-Nanopartikel. Dementsprechend müssen wir mit einer gewissen chromosomalen Integration der kontaminierenden Plasmid-DNA in vivo rechnen.
4.4. Entzündungsfördernde Wirkung der bakteriellen DNA
Das angeborene Immunsystem des Menschen reagiert mit Entzündungen auf verschiedene bakterielle Makromoleküle, darunter auch DNA. Es ist davon auszugehen, dass die in den Impfstoffen enthaltenen großen Mengen an DNA zu Entzündungen in der Nähe der Injektionsstelle und möglicherweise auch an anderen Stellen im Körper beitragen.
5. Schlussfolgerung
Das Vorhandensein von kontaminierender Plasmid-DNA in den mRNA-Impfstoffen von Pfizer und Moderna birgt schwerwiegende Gesundheitsrisiken, die zu den bereits bekannten und verstandenen Risiken hinzukommen. Zu diesen Risiken gehören vor allem die verlängerte Expression des Spike-Proteins, die zu einer entsprechend verlängerten und zerstörerischeren autoimmunähnlichen Entzündung führen kann, sowie die Induktion bösartiger Erkrankungen nach chromosomaler Integration der Plasmid-DNA. Darüber hinaus beweist das schiere Ausmaß der Kontamination eindeutig, dass die Hersteller die vorgesehenen Produktionsverfahren nicht beherrschen oder nicht ordnungsgemäß umgesetzt haben. Jeder dieser Punkte allein wäre Grund genug, die sofortige Rücknahme dieser Impfstoffe zu fordern.
Danksagungen
Wir danken Kevin McKernan und Ulrike Kämmerer für Korrekturen und Diskussionen.
Urheberrecht
Dieser Text ist lizenziert unter den Bedingungen der Creative Commons Attribution 4.0 International License (CC BY 4.0). Das bedeutet, dass Sie den Inhalt frei kopieren und wiederverwenden können, solange die ursprünglichen Autoren genannt werden. Wenn Sie Änderungen am Text vornehmen, müssen Sie dies ausdrücklich angeben. Weitere Informationen finden Sie auf der Creative-Commons-Website[43].
Quellenangaben
- Andries, O. et al. (2015) N1-Methylpseudouridin-inkorporierte mRNA übertrifft pseudouridin-{}inkorporierte mRNA durch verbesserte Proteinexpression und reduzierte Immunogenität in Säugetierzelllinien und Mäusen. J. Control.Release 217:337-344
- Anonym, (2020) FDA-Briefing-Dokument: Pfizer-BioNTech COVID-19-Impfstoff.
- Anonym, (2020) FDA-Briefing-Dokument: Moderna MRNA-1273.
- Anonym, (2021) EMA Bewertungsbericht: Komirnaty.
- Anonym, (2021) EMA-Bewertungsbericht: COVID-19-Impfstoff Moderna.
- McKernan, K. (2023) Deep sequencing of the Moderna and Pfizer bivalent vaccines identifies contamination of expression vectors designed for plasmid amplification in bacteria.
- McKernan, K. (2023) Die bivalenten Impfstoffe von Pfizer und Moderna enthalten 20-35% Expressionsvektor und sind in E.coli transformationsfähig.
- McKernan, K. (2023) DNA-Kontamination in 8 Fläschchen mit monovalenten mRNA-Impfstoffen von Pfizer.
- Patel, H.K. et al. (2023) Charakterisierung von BNT162b2 mRNA zur Bewertung des Risikos einer Off-Target-Antigen-Translation. J. Pharm.Sci. DOI:10.1016/j.xphs.2023.01.007
- Pardi, N. et al. (2018) Nukleosid-modifizierte mRNA-Impfstoffe induzieren starke T-follikuläre Helfer- und Keimzentrums-B-Zellantworten. J. Exp. Med. 215:1571-1588
- Bansal, S. et al. (2021) Cutting Edge: Circulating Exosomes with COVID Spike Protein Are Induced by BNT162b2 (Pfizer-BioNTech) Vaccination prior to Development of Antibodies: Ein neuer Mechanismus zur Aktivierung des Immunsystems durch mRNA-Impfstoffe. J. Immunol. 207:2405-2410
- Castruita, J.A.S. et al. (2023) SARS-CoV-2 spike RNA vaccine sequences circulate in blood up to 28 days after COVID-19 vaccination. APMIS 131:128-132
- Fertig, T.E. et al. (2022) Impfstoff-mRNA kann bis zu 15 Tage nach der Impfung im Blut nachgewiesen werden. Biomedizin 10:1538
- Magen, E. et al. (2022) Klinische und molekulare Charakterisierung eines seltenen Falls von BNT162b2 mRNA COVID-19-Impfstoff-assoziierter Myositis. Impfstoffe 10
- Röltgen, K. et al. (2022) Immunprägung, Breite der Variantenerkennung und Keimzentrumsantwort bei menschlicher SARS-CoV-2-Infektion und Impfung. Cell DOI:10.1016/j.cell.2022.01.018
- Miao, C.H. et al. (2001) Langfristige und therapeutisch wirksame hepatische Genexpression des menschlichen Faktors IX nach nacktem Plasmidtransfer in vivo. Mol. Ther. 3:947-57
- Ye, X. et al. (2003) Vollständige und anhaltende phänotypische Korrektur der Hämophilie B bei Mäusen nach hepatischem Gentransfer eines hochexprimierenden menschlichen Faktor IX-Plasmids. J. Thromb. Haemost. 1:103-11
- Jager, L. und Ehrhardt, A. (2009) Persistenz von adenoviralen Vektoren mit hoher Kapazität als replikationsdefekte monomere Genome in vitro und in der Leber von Mäusen. Hum. Gene Ther. 20:883-96
- Li, Y.Q. et al. (2000) [Die Funktion des T7-Promotors als cis-wirkendes Element für die Polymerase II in eukaryotischen Zellen]. Yi Chuan Xue Bao 27:455-61
- Mörz, M. (2022) Ein Fallbericht: Multifokale nekrotisierende Enzephalitis und Myokarditis nach BNT162b2 mRNA-Impfung gegen Covid-19. Vaccines 10:2022060308
- Byrne, B.J. et al. (1983) Definition der frühen Promotorregion des Affenvirus 40 und Nachweis einer Wirtsbereichsbias in der Verstärkungswirkung der 72-Basen-Paar-Wiederholung des Affenvirus 40. Proc. Natl. Acad.Sci. U. S. A. 80:721-5
- Rotondo, J.C. et al. (2019) Assoziation zwischen Simian Virus 40 und menschlichen Tumoren. Front. Oncol. 9:670
- DeCaprio, J.A. und Garcea, R.L. (2013) A cornucopia of human polyomaviruses. Nat. Rev. Microbiol. 11:264-76
- Hussain, I. et al. (2020) Human BK and JC polyomaviruses: Molekulare Erkenntnisse und Prävalenz in Asien. Virus Res. 278:197860
- La Bella, F. und Ozer, H.L. (1985) Differentielle Replikation von SV40- und Polyoma-DNAs in Ovarialzellen des chinesischen Hamsters. Virus Res. 2:329-44
- Ehrhardt, A. et al. (2003) Episomale Persistenz von rekombinanten adenoviralen Vektorgenomen während des Zellzyklus in vivo. J. Virol. 77:7689-95
- Baltimore, D. (1970) RNA-abhängige DNA-Polymerase in Virionen von RNA-Tumorviren. Natur 226:1209-11
- Doerfler, W. (2016) Vorsicht vor Manipulationen am Genom: Epigenetische Destabilisierung durch (fremde) DNA-Insertionen. Epigenomics 8:587-91
- Doerfler, W. (1996) Ein neues Konzept in der (adenoviralen) Onkogenese: Integration von Fremd-DNA und ihre Folgen. Biochim. Biophys.Acta 1288:F79-99
- Southern, P.J. und Berg, P. (1982) Transformation von Säugetierzellen zur Antibiotikaresistenz mit einem bakteriellen Gen unter Kontrolle des SV40 early region promoter. J. Mol. Appl. Genet. 1:327-41
- Stuchbury, G. und Münch, G. (2010) Optimierung der Erzeugung stabiler neuronaler Zelllinien durch Restriktionsenzymverdau vor der Transfektion von Plasmid-DNA. Cytotechnology 62:189-94
- Aldén, M. et al. (2022) Intrazelluläre Reverse Transkription des Pfizer BioNTech COVID-19 mRNA-Impfstoffs BNT162b2 in vitro in einer menschlichen Leberzelllinie. Curr. Issues Mol.Biol. 44:1115-1126
- Zhang, L. et al. (2020) SARS-CoV-2 RNA revers-transkribiert und in das menschliche Genom integriert. bioRxiv DOI:10.1101/2020.12.12.422516
- Staal, F.J.T. et al. (2008) Sola dosis facit venenum. Leukämie in Gentherapieversuchen: eine Frage von Vektoren, Inserts und Dosierung? Leukämie 22:1849-1852
- Kohn, D.B. und Gaspar, H.B. (2017) How We Manage Adenosine Deaminase-Deficient Severe Combined Immune Deficiency (ADA SCID). J. Clin. Immunol. -:n/a
- Doerfler, W. et al. (2018) Inheritable epigenetic response towards foreign DNA entry by mammalian host cells: a guardian of genomic stability. Epigenetics 13:1141-1153
- Sipehia, R. und Martucci, G. (1995) Hocheffiziente Transformation von humanen Endothelzellen durch Apo E-vermittelte Transfektion mit Plasmid-DNA. Biochem. Biophys. Res. Commun. 214:206-11
- Takahashi, M. et al. (2002) Transformation von MC3T3-E1-Zellen nach Stress und Transfektion mit pSV2neo-Plasmid. Anticancer Res. 22:585-98
- Laurema, A. et al. (2003) Transfektion von Eizellen und anderen Arten von Eierstockzellen bei Kaninchen nach direkter Injektion von Adenoviren und Plasmid/Liposomen in die Gebärmutterarterien. Gene Ther. 10:580-4
- Dhup, S. und Majumdar, S.S. (2008) Transgenese durch permanente Integration von Genen in repopulierende Spermatogonienzellen in vivo. Nat.Methoden 5:601-3
- Anonym, (2020) Long Term Follow-Up After Administration of Human Gene Therapy Products: Leitfaden für die Industrie.
- Wang, Z. et al. (2004) Nachweis der Integration von Plasmid-DNA in genomische Wirts-DNA nach intramuskulärer Injektion und Elektroporation. Gene Ther. 11:711-21
- Anonym, (2023) Creative Commons Namensnennung 4.0 Internationale Lizenz (CC BY 4.0).
Ursprünglich veröffentlicht auf Doctors for Covid Ethics
Korrektur vorschlagen