Pruebas sorprendentes sugieren que BioNTech/Pfizer falsificó datos clave y participó en otros escándalos (2ª parte)

Publicado originalmente en Trial Site News
Parte 1 de mi reportaje de investigación publicado en Trial Site News se centraba en los «Western Blots» de Pfizer-BioNTech, de aspecto inusual, el escándalo conocido como «Blotgate». Los Western Blots fueron realizados por BioNTech para demostrar la fidelidad de su producto (a los reguladores) que sólo la esperada proteína spike estaba siendo expresada in vitro por el ARNm vacunal modificado y era consistente a través de diferentes lotes.
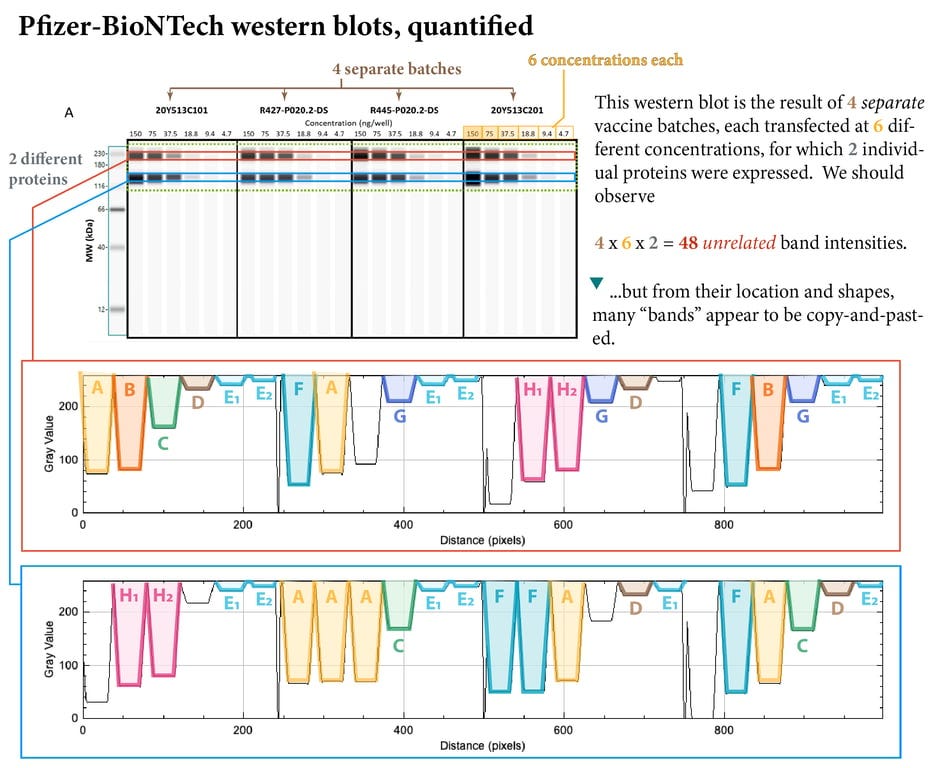
El problema de que no parezcan Western blots auténticos/convencionales, sino versiones generadas por ordenador (Western blots automatizados, a pesar de no estar etiquetados como tales en ninguno de los informes), es una distracción del verdadero escándalo que nos ocupa: las pruebas, que muestran prima facie que BioNTech/Pfizer «copiaron y pegaron» estas bandas, en otras palabras, falsificaron sus datos clave. Estas bandas occidentales aparentemente fabricadas (que se muestran a continuación) se exhibieron en la respuesta de Pfizer a las consultas de la FDA » Administración de Alimentos y Medicamentos de EE.UU» alrededor de noviembre de 2020, en el período previo a la Autorización de Uso de Emergencia.
Sólo cuando estas bandas se cuantificaron mediante análisis de software de imagen (gracias a un experto anónimo), el trabajo de «copiar y pegar», realizado a través de 4 lotes separados de su producto, transfectados a 6 concentraciones diferentes, se hizo sorprendentemente visible.
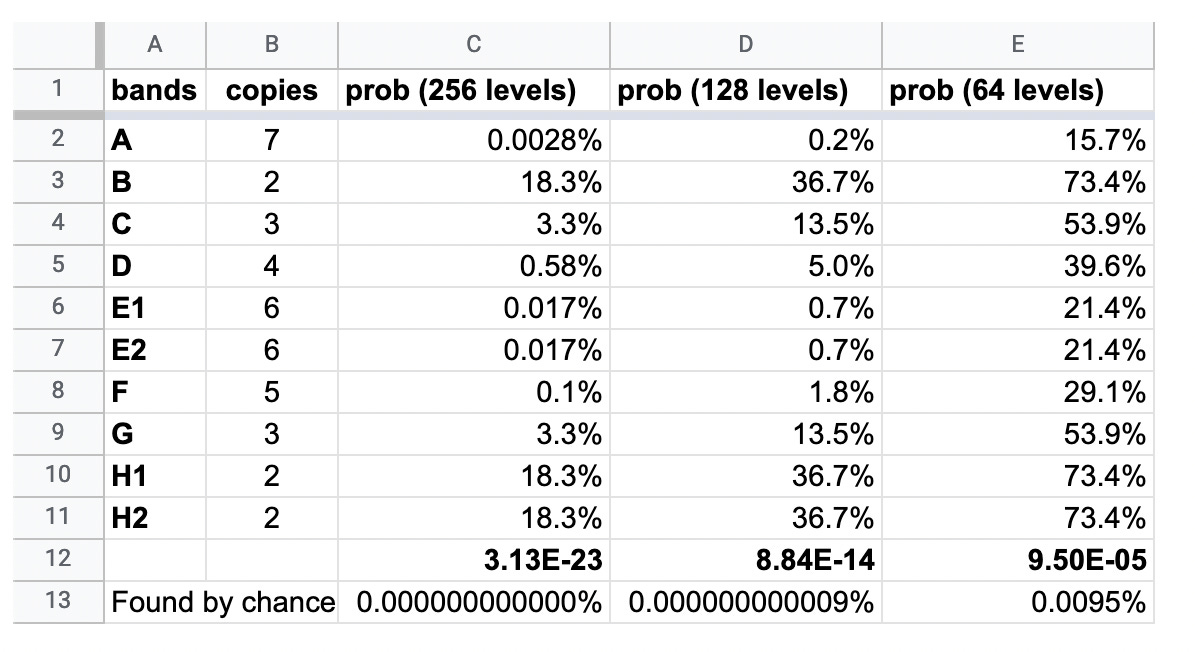
Las probabilidades de que aparezcan exactamente las mismas bandas de forma natural en cuatro lotes distintos de la vacuna, transfectados a 6 concentraciones diferentes, se pueden ver en la siguiente tabla.
A raíz de la Parte 1 de mi informe de investigación, Epoch Times llevó a cabo su propia investigación sobre Blotgate; los problemas de calidad del ARNm de la vacuna de Pfizer-BioNTech y se refirió a la investigación de Trial Site News sobre la filtración de correos electrónicos y documentos de la Agencia Europea de Medicamentos (EMA).
«Escondiendo los trapos sucios»: una mirada más al Blotgate
El 13 de enero, poco después de que estallara el escándalo Blotgate, el artículo patrocinado por Pfizer y BioNTech, Patel et al. ‘Characterization of BNT162b2 mRNA to Evaluate Risk of Off-Target Antigen Translation‘ fue publicado en el Journal of Pharmaceutical Sciences. El artículo presenta muchas anomalías y merece la pena señalar que la Dra. Jessica Rose, bióloga computacional y molecular, ha escrito un exhaustivo análisis crítico de este estudio.
En primer lugar, cabe destacar los intereses contrapuestos de los autores, que pueden verse a continuación.

Al hablar con la Dra. Jessica Rose, que ha realizado ampliamente Western Blots convencionales, explicó: «En mi opinión experta, [un Western tradicional] es uno de los ensayos de banco que es un procedimiento, como las pruebas de seguridad para biológicos (que se supone que duran 10 años). Un western blot requiere una secuencia de pasos específicos, estos pasos no pueden apresurarse ni solaparse… los experimentos realizados con Robo Jess [máquina automatizada de Western Blot] y los blots presentados por Pfizer deben ser reproducidos por manos humanas, la reproducibilidad es un requisito, especialmente cuando la lista de conflictos de intereses es tan larga por parte de los autores remitentes [Patel et al.]»
La siguiente imagen, tomada del artículo de Patel et al. muestra su «Western Blot» Obsérvense las bandas negras regulares muy gruesas (homogéneas) sin ningún tipo de mancha.
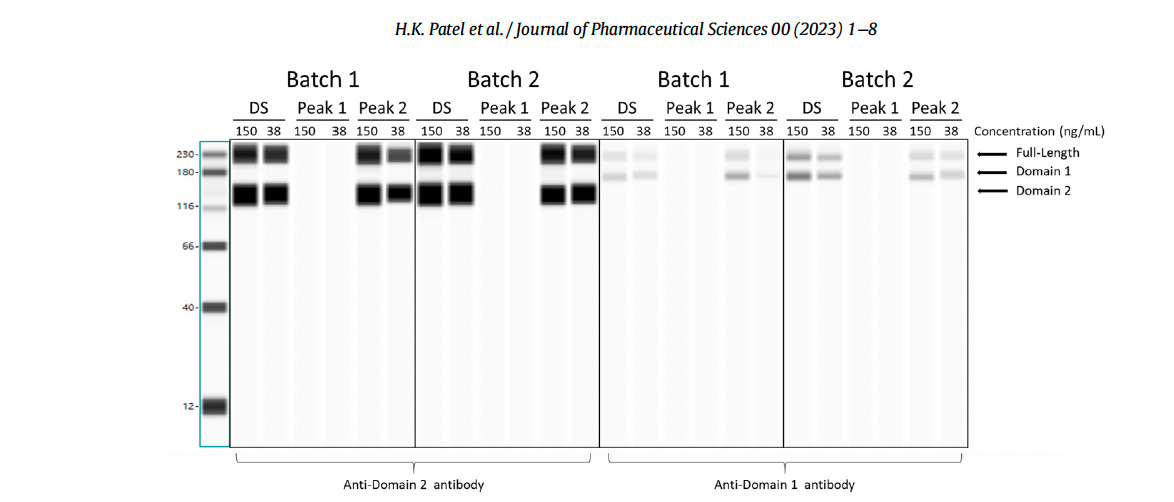
El mismo «Western Blot» (que se ve a continuación), pero en una versión mucho más burda y escaneada, se puede encontrar en el informe redactado del CHMP (Comité de Medicamentos de Uso Humano) de la Agencia Europea de Medicamentos de agosto de 2021, aproximadamente 18 meses antes del artículo de Patel et al.
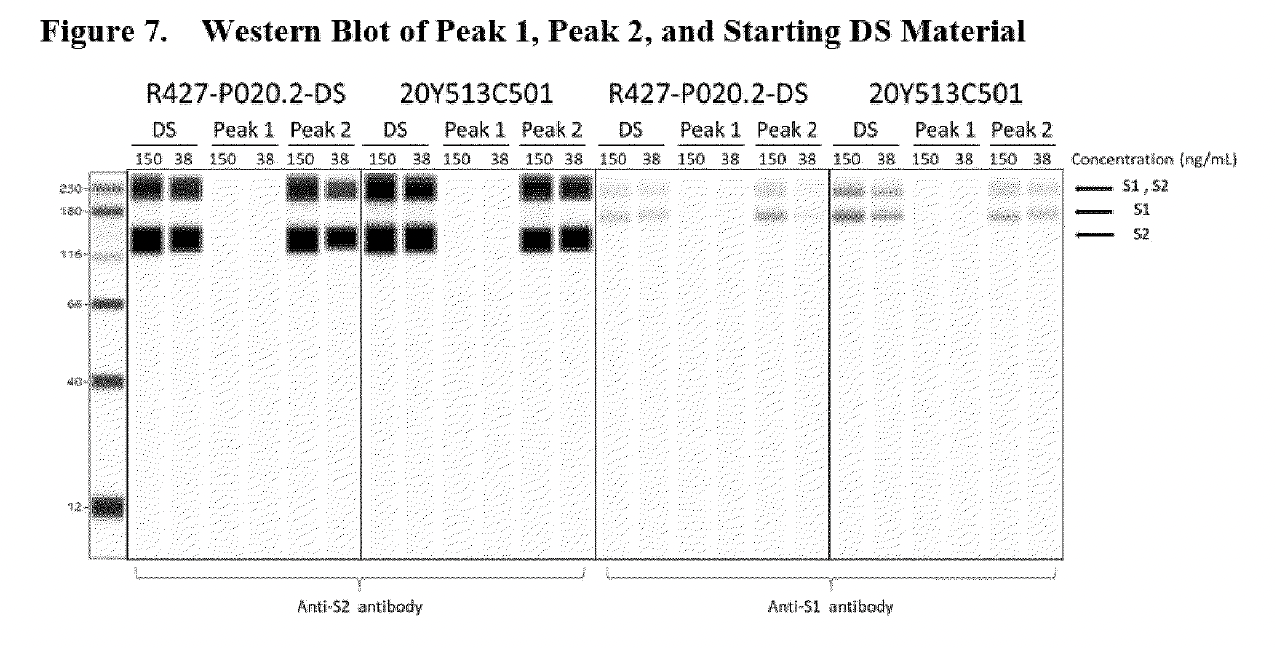
Dado que los autores de Patel et al. hacen referencia a la tecnología de ProteinSimple en su estudio (extracto a continuación), se puede hacer una comparación con una muestra de un Western Blot automatizado, utilizando el software de la misma empresa.
‘Los lisados celulares se analizaron con los anticuerpos específicos para detectar las proteínas de la espiga SARS-CoV-2 utilizando la tecnología ProteinSimple. se utilizó el módulo de separación Wes de 12-230 kDa y 25 cartuchos capilares. Se utilizaron el anticuerpo de ratón SARS-CoV-2 Spike S1 subunit (R&D systems, Nº de catálogo MAB105403) y el anticuerpo de ratón SARS-CoV-2 Spike S2 subunit (R&D systems, Nº de catálogo MAB10557). Los resultados de las muestras de ARNm se presentan como imágenes de carril de ejecución del ensayo ProteinSimple Wes.’
Fuente: Patel et al. paper
La imagen de abajo es de un Western automatizado utilizando la tecnología de ProteinSimple.
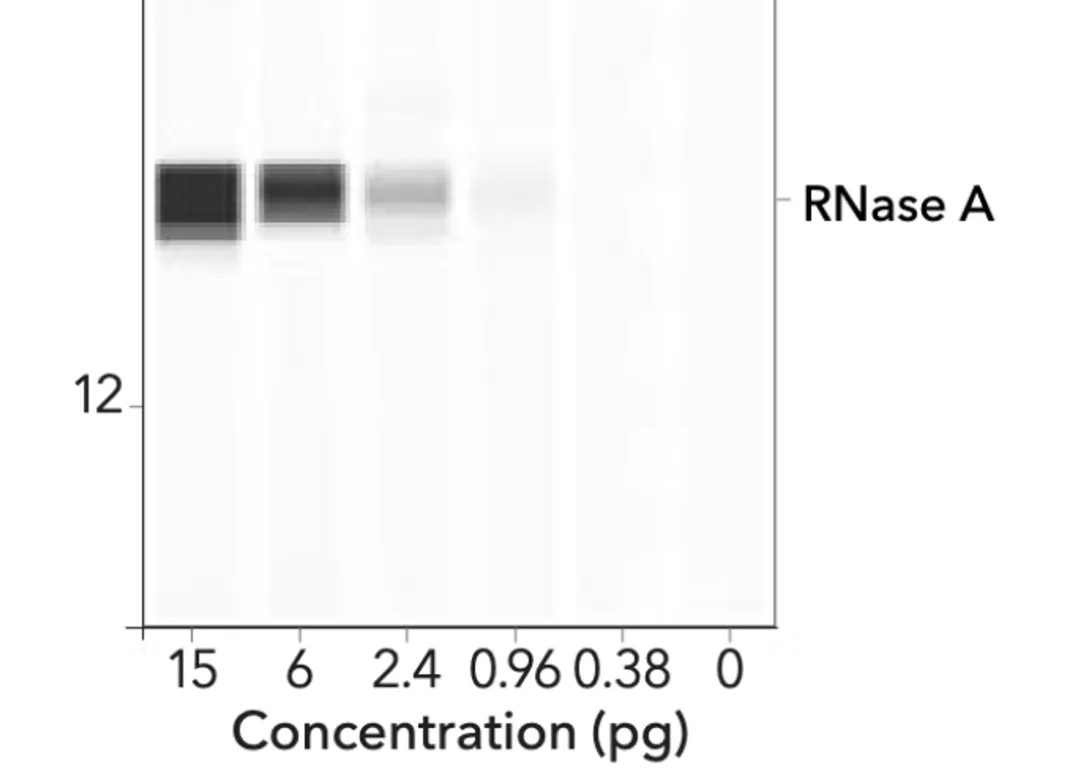
Lo que llama la atención es que puede observarse un gradiente (efecto difuminado) en cada una de las bandas variadas. Su aspecto es muy diferente al de las bandas negras, gruesas y homogéneas de BioNTech/Pfizer.
Para obtener más información, entrevisté al director de I+D del Proyecto Genoma Humano y científico genómico, Kevin McKernan, quien explicó que «los datos de BioNTech/Pfizer son inutilizables» y que su razonamiento para el aspecto inusual de las bandas de Western Blot (que se ven en el informe de la EMA y en el artículo de Patel) se debe a que «ellos [BioNTech] aumentaron mucho la ganancia y la mayoría de la gente hace eso para ocultar las ‘bandas sucias’ Probablemente haya 5 bandas ahí [en una banda gruesa de aspecto único]» Me refiero a esa entrevista informativa, más adelante en este informe.
Esto nos lleva a preguntarnos si BioNTech/Pfizer encubrieron intencionadamente los resultados de sus «Western blot» presentando versiones manipuladas (de Westerns automatizados) a las autoridades reguladoras ¿Quizás un trabajo de copiar y pegar para la FDA y una manipulación de los niveles de saturación para la EMA? Y lo que es más importante: ¿cómo aceptaron los reguladores estos «Western blots» como prueba principal de la fidelidad y consistencia del producto de BioNTech y Pfizer?
Me puse en contacto con la EMA para obtener una respuesta a las inquietudes planteadas en esta investigación. Su responsable de prensa respondió con la siguiente declaración: «Estas cifras [imágenes de Western Blot] se extrajeron del expediente presentado y se insertaron en el informe de evaluación, lo que provocó una pérdida de calidad de la imagen. Además, el software de redacción utilizado por la EMA para preparar los documentos para su publicación tras una solicitud de DTA tiene un impacto en la resolución de los documentos.
Con respecto a la validación del método, los métodos analíticos Western Blot utilizados en los estudios de caracterización se evaluaron previamente durante la solicitud de autorización de comercialización condicional inicial como parte de la «evaluación del historial de desarrollo y comparabilidad» y se consideraron adecuados para ese fin»
La FDA, Pfizer y BioNTech no respondieron para hacer comentarios.
Parámetros de calidad fijados meses después de la concesión de la ACM
La razón por la que el titular de la autorización de comercialización (BioNTech) llevó a cabo una serie adicional de pruebas fue porque la autorización condicional de comercialización (ACM) se concedió sobre la base de que el titular de la ACM cumpliera con las obligaciones específicas (tales como datos adicionales para caracterizar aún más las especies de ARNm truncado y modificado) que le impuso la EMA. Hasta el momento en que se concedió la ACM, la EMA señaló múltiples problemas de CMC (Química, Fabricación y Controles) como principales preocupaciones, en particular la caída en la integridad del ARNm (presencia de ARNm truncado/fragmentado – carente de un atributo crítico, una 5’cap y/o cola poli (A)) de los lotes comerciales en comparación con los utilizados en los ensayos clínicos. La «solución» a este grave problema fue rebajar los criterios de aceptación de las especies de ARNm fragmentado/truncado al 50%, algo que los reguladores simplemente dejaron pasar.
La farmacéutica canadiense Maria Gutschi, PharmD, que cuenta con más de 30 años de experiencia en el sector hospitalario, comunitario y gubernamental, analizó de forma independiente los problemas de calidad de la vacuna de Pfizer/BioNTech identificados por la Agencia Europea del Medicamento, en un vídeo de presentación informativo. Al hablar con Gutschi, planteó otras preocupaciones importantes: no fue hasta mayo de 2021 [5 meses después de concederse la autorización] cuando la OCABR estableció los parámetros de calidad y, lo que es más importante, la normalización de los ensayos utilizados. Un problema que encontré fue que en el lanzamiento, muchos de los ensayos utilizados para determinar la calidad y la identidad eran ensayos «internos» de BioNTech. Pues bien, un regulador no puede aceptar eso sin más. Hay que validarlos para que sean coherentes, reproducibles y fiables«
OCABR son las siglas de Official Control Authority for Batch Release, que establece las directrices para las vacunas humanas autorizadas en la UE. Fíjese en la fecha resaltada de mayo de 2021 del documento de la OCABR, que se ve a continuación.

El hecho de que la OCABR estableciera los primeros parámetros de calidad de una vacuna humana, 5 meses después de que se concediera la autorización oficial de comercialización, no tiene precedentes. En segundo lugar, el hecho de que estos ensayos (pruebas) tuvieran que ser validados es digno de mención.
Los cuestionables ensayos «internos«
Vale la pena señalar que la preocupación de Gutschi con los ensayos «internos» de BioNTech también fue compartida por un evaluador de la EMA en el Informe de Revisión Rodante del Relator filtrado de noviembre de 2020, poco antes de que se concediera la CMA.

Volviendo al documento de orientación de la FDA de 2016 sobre «Integridad de los datos y cumplimiento de las CGMP», en él se afirma: ‘En los últimos años, la FDA ha observado cada vez más infracciones de las CGMP relacionadas con la integridad de los datos durante las inspecciones de las CGMP. Esto es preocupante porque garantizar la integridad de los datos es un componente importante de la responsabilidad de la industria para garantizar la seguridad, eficacia y calidad de los medicamentos, y de la capacidad de la FDA para proteger la salud pública.’
La consideración por parte de la FDA de las violaciones de la integridad de los datos como «preocupantes» parece desvanecerse al aceptar de buen grado los datos aparentemente «copiados y pegados» de BioNTech. Tal vez, era sólo un ejercicio de marcar la casilla para ellos, para aparecer como si estuvieran haciendo su debida diligencia en la «protección de la salud pública»
Las bandas «inesperadas» que aparecen en los Western blots auténticos de BioNTech
Entre todos los «Western blots» generados por ordenador, BioNTech presentó dos Western auténticos (el segundo se analizará más adelante), lo que demuestra que sabían cómo hacerlos. En el informe filtrado de evaluación de la Revisión Rodante del Relator de la EMA, se hacían las siguientes críticas al estudio 20-0211 de BioNTech ‘en relación con los resultados obtenidos del Western Blot’, que se señalan a continuación.

El pasado mes de febrero, el estudio 20-0211 (el estudio especificado anteriormente) realizado por BioNTech fue publicado como parte del volcado de datos de Pfizer ordenado por el tribunal. El Western Blot auténtico altamente revelador exhibido en ese estudio se puede ver a continuación.

Del informe de evaluación de la EMA de noviembre de 2020, sabemos que la agencia señaló los pesos moleculares inesperados (medidos en kDa) de las dos bandas de proteínas mostradas en la prueba Western Blot, en 190kDa y 100 kDa respectivamente, lo que les llevó a exigir una explicación de BioNTech. Ahora bien, la proteína de la espiga completa tiene un peso molecular de 141 kDa y la S1 (subunidad de la proteína de la espiga) tiene un peso molecular de 76,5 kDa, que se utilizó como control. Obsérvese, en la figura anterior, que no se muestran bandas de proteínas con ninguno de los pesos moleculares esperados en el carril BNT 162B2 y que el peso molecular esperado de 76,5 kDa de la proteína S1 no se observó en el carril de control S1. Estas anomalías llamaron la atención del regulador, pero de alguna manera el desarrollador de la vacuna, BioNTech, que seguramente habría conocido el peso molecular esperado de la proteína espiga completa, ya que se supone que su producto ARNm la codifica, ni siquiera las reconoció.
Para agravar su completo fracaso a la hora de abordar estos problemas, BioNTech escribió una descripción muy inexacta de su Western Blot – de hecho, exactamente lo contrario de lo que se mostraba en la imagen: bNT162b2 tiene un tamaño previsto de 141,14 kDa» y «la proteína de la subunidad S1 del SARS-CoV-2 (76,5kDa) se utilizó como control positivo»
Esto sugiere que tal vez la proteína de la subunidad S1S2 no haya sido expresada por el ARNm modificado de la vacuna o, si lo fue, tal vez también se expresaran otras proteínas aberrantes, derivadas de moléculas de ARN fragmentadas/truncadas en la sustancia farmacológica.
Ausencia de secuenciación del genoma y problemas con la metilpseudourina N1
Cuando se le preguntó sobre los problemas relacionados con la calidad de la vacuna de Pfizer-BioNTech, el científico genómico Kevin McKernan dio la siguiente respuesta:
No tenemos ninguna secuencia de ADN en estos lotes. ¡Es una locura! Viniendo del proyecto del genoma humano, publicabamos una secuencia cada 24 horas para asegurarnos de que el mundo tuviera acceso a los datos más recientes del proyecto del genoma humano. Ahora no hay ninguna secuencia del genoma de los lotes«
Es asombroso que los reguladores se basaran en estos Westerns blots de aspecto manipulado de BioNTech, en lugar de solicitar secuencias genómicas de los lotes para demostrar su coherencia y fidelidad».
McKernan continuó refiriéndose a los problemas causados por el ARN modificado sintetizado utilizando una base modificada: «Tenemos un producto de ARNm en el que cada uridina ha sido sustituida por N1 Metilpseudourina, que el cuerpo nunca ha visto antes. Ellos [BioNTech y Pfizer] eligieron codones de parada que son los más conocidos por crear errores. Conocían el problema, pero no lo corrigieron adecuadamente. Esto significa que cuando los ribosomas van a leer la plantilla, están realmente confundidos, ya que nunca la habían visto antes»
McKernan coescribió un artículo con el Dr. Peter McCullough y Anthony Kyriakopoulos titulado «Differences in Vaccine and SARS-CoV-2 Replication Derived mRNA: Implications for Cell Biology and Future Disease» Los autores concluyeron que «los cambios de codones sinónimos incorporados a las vacunas de ARNm pueden alterar la conformación esperada de la proteína codificada, ya que la velocidad y la eficiencia de la traducción pueden dar lugar a un plegamiento de la proteína diferente… Las estrategias de optimización de codones para el desarrollo de vacunas de ARNm pueden dar lugar a desregularidades inmunológicas, afectar a la regulación epi-transcriptómica y conducir a la progresión de la enfermedad»
La uridina modificada (N1 Metilpseudorina) se incorporó al ARNm para eludir la respuesta inmunitaria innata y promover la traducción de proteínas. Sin embargo, en el informe Rolling Review de la EMA de noviembre de 2020 (página 61), se planteó un riesgo potencial de seguridad con el ARN modificado (modARN), véase más abajo.
el modARN contiene una sustitución de 1-metil-pseudouridina por uridina. Esta sustitución disminuye el reconocimiento del ARN de la vacuna por parte de los sensores inmunitarios innatos. Sin embargo, no se proporcionó más información sobre el riesgo de respuestas autoinmunes inducidas por el modARN. Se invita al Solicitante a discutir más a fondo la posibilidad de que la vacuna de ARNm pueda desencadenar posibles respuestas autoinmunes y cómo planea evaluar posiblemente su aparición»
Se desconoce si BioNTech evaluó alguna vez el riesgo potencial de seguridad de la respuesta autoinmune causada por la uridina modificada o las proteínas traducidas (aparte de la proteína spike) porque en agosto de 2021, con la fecha límite de julio de 2021 ya pasada, todavía no se había hecho. (Véase la instantánea a continuación, página 13 del informe de agosto de 2021 del CHMP de la EMA)

Lo que resulta inquietante son los resultados del mayor estudio de este tipo del Hospital Universitario Rey Fahad de Khobar (Arabia Saudí), que vincula las vacunas de ARNm con el desencadenamiento de enfermedades autoinmunes, que TrialSite informó recientemente. el «mimetismo molecular», mencionado como motivo de preocupación en el informe de la EMA, es la misma hipótesis planteada en el estudio que el mecamismo asociado con las vacunas de ARNm que causan un proceso autoinmune.
Posibles problemas derivados del proceso de fabricación
La siguiente figura muestra la visión simplista utilizada para promover el funcionamiento de las vacunas de ARNm.
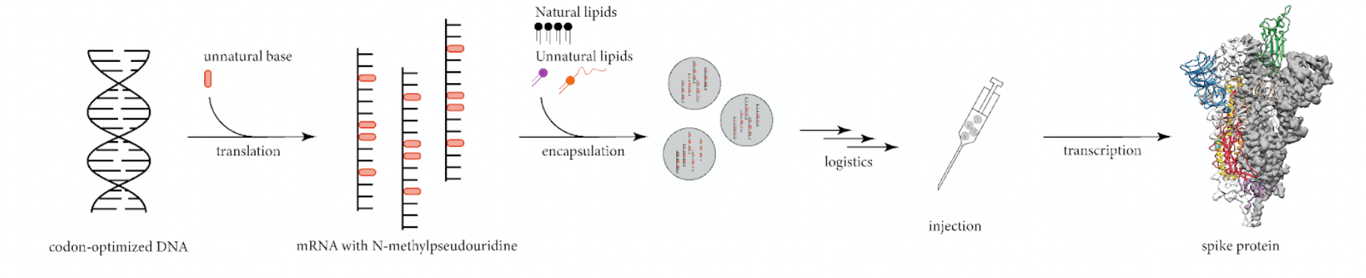
Cuando, en realidad, el proceso es muy variable, lo que a su vez genera productos proteicos muy variables, véase la figura siguiente.

Cada paso del proceso de fabricación puede introducir impurezas desconocidas y errores desconocidos, desde la traducción «infiel» del ADN optimizado en codones, que puede generar fragmentos de ARNm (no intactos), hasta la transcripción «infiel» de la mezcla de ARNm en el cuerpo humano, que puede generar productos proteínicos inesperados.
McKernan continuó explicando la tasa de error y sus implicaciones: según mis cálculos, hay un error en cada molécula de la vacuna -teniendo en cuenta que hay 14 billones de moléculas en cada inyección-, pero no sabemos lo que eso significa desde el punto de vista inmunológico. Por eso hay que secuenciar mucho antes de inyectar en humanos un producto que se sabe que produce proteínas nuevas«
El ARNm modificado de longitud completa de la vacuna de Pfizer-BioNTech que codifica la proteína espiga tiene ∼4300 nucleótidos, nt de longitud. Todo lo que sea menos que eso se considera una especie de ARNm fragmentado, que la EMA clasificó como «impureza relacionada con el producto». Esto lleva a otro escándalo más, el conocido como Humpgate.
El escándalo Humpgate
El Humpgate puede considerarse el precursor del Blotgate. Mientras que el Blotgate se centraba en las bandas proteínicas de aspecto falso expresadas por el ARNm vacunal observado en los Westerns de BioNTech, el Humpgate se refiere a las especies de ARNm truncado señaladas por la EMA y otros organismos reguladores. El nombre procede de las «jorobas» que se ven en la imagen de la publicación en las redes sociales que figura a continuación.
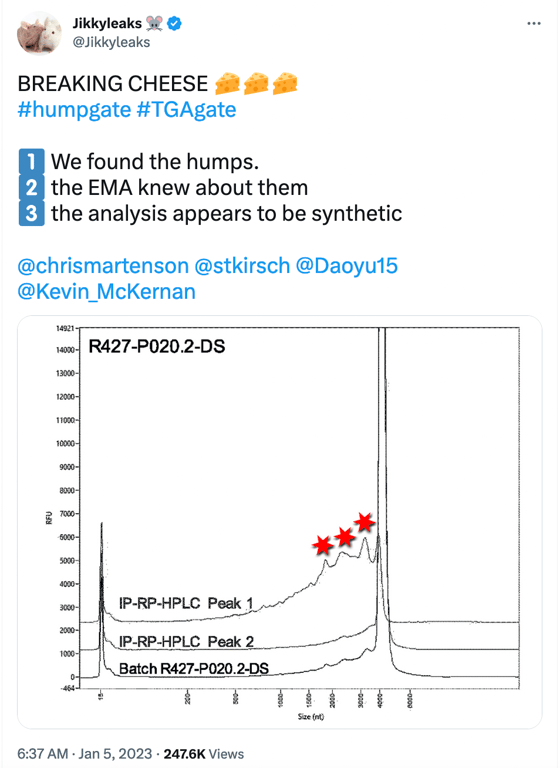
Las 3 «jorobas» marcadas con estrellas rojas representan las especies de ARN truncado/fragmentado con una longitud acortada (nt) <4000nt. La imagen de arriba es de un electroferograma del Analizador de Fragmentos (que es la imagen superior de la Figura 2 que se muestra a continuación) tomada de la página 15 del informe redactado de la EMA del verano de 2021, que BioNTech llevó a cabo con el fin de cumplir con la SO1 (obligación específica 1) establecida por la EMA, que solicitó datos adicionales para caracterizar con más detalle las especies de ARNm truncadas y modificadas.

Se tomaron muestras fraccionadas del Proceso 1(R427-P020.2-DS, lote de ensayo clínico, imagen superior) y del Proceso 2(20Y513C501, lote PPQ, imagen inferior) utilizando RP-HPLC de emparejamiento iónico para caracterizar más las especies de ARNm intacto (de longitud completa) y fragmentado. Ambos lotes (Proceso 1 y 2) muestran pequeñas jorobas que conducen a un gran pico de alrededor de 4300 nt de tamaño, que representa la especie de ARNm de longitud completa. La línea del gráfico etiquetada como Pico 1 muestra el material «purificado» reanalizado compuesto sólo por las especies de ARNm fragmentado (son las pequeñas jorobas que se ven a la izquierda del pico grande de unos 4300 nt de la muestra del lote) y el Pico 2 es el material «purificado» reanalizado compuesto por las especies de ARNm intacto, que se ve en el «pico grande»
Este mismo electroferograma se puede encontrar en el artículo de Patel et al. publicado en enero de 2023 (véase la imagen de abajo, publicada 18 meses después del informe de la EMA). Sin embargo, los autores de Patel et al. citan su investigación como actual cuando es una versión reelaborada de la propia respuesta de Pfizer/BioNTech a las preguntas de la FDA y la EMA de hace 18 meses. El hecho de que los autores hayan presentado su estudio como «independiente» y «actual», cuando la tecnología ProteinSimple Wes utilizada en su investigación fue descontinuada a partir del 30 de julio de 2021, es cuando menos falso.
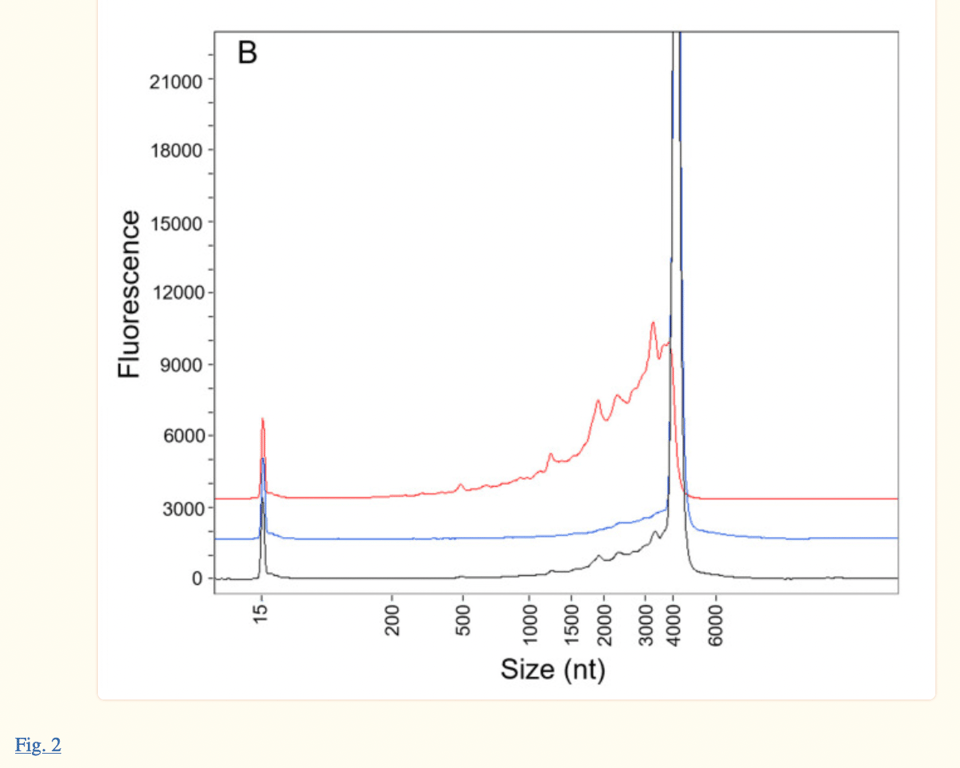
Tanto el artículo de Patel et al. como el documento de la EMA restan importancia a las jorobas observadas en el lado izquierdo del pico principal en torno a 4300 nt. En el informe de la EMA se afirma que «(Figura 2) demuestra que el pico 1 consiste casi en su totalidad de especies fragmentadas, en consonancia con los datos proporcionados anteriormente en la Evaluación de la Respuesta al CHMP Q01-Calidad 11-Dic-2020»
El problema con la hipótesis de BioNTech
La forma en que BioNTech aseguró a la EMA que estas especies truncadas de ARN no serían capaces de apoyar la traducción de proteínas, por lo que las especies truncadas vistas como ‘jorobas’ en los electroferogramas no se verían como un problema, fue mediante la realización de nuevos ensayos Western Blot para demostrar que el ARN ‘transcripciones requieren tanto 5′-cap y poli (A) para apoyar la traducción de proteínas ..’

Este es el aspecto de un ARN de longitud completa BNT162b2. Al principio del transcrito de ARN se añade una tapa de 5′ y al final una cola de poli-A de 3′. Los ribosomas leen el transcrito de 5′ a 3′.

Las siguientes figuras (6 y 5) proceden del informe CHMP de la EMA de agosto de 2021.

Los carriles en blanco exhibidos en los paneles de Sustancia Fármaco No Poly(A), fueron suficientes para probar al regulador que el ARN que carece de un Poly (A) no sería capaz de expresar la proteína pico S1S2. Sin embargo, el problema de este ensayo es que no confirma que cualquier otra proteína aberrante (que era una preocupación inicial de la EMA) pudiera ser expresada por este ARN truncado, ya que sólo se utilizaron los anticuerpos de detección específicos para el dominio S1 y S2. El Western Blot que se muestra a continuación comparte el mismo problema. Este ensayo se utilizó para asegurar al regulador que el transcrito de ARN que carece de un 5’cap no sería capaz de expresar la proteína pico S1S2.
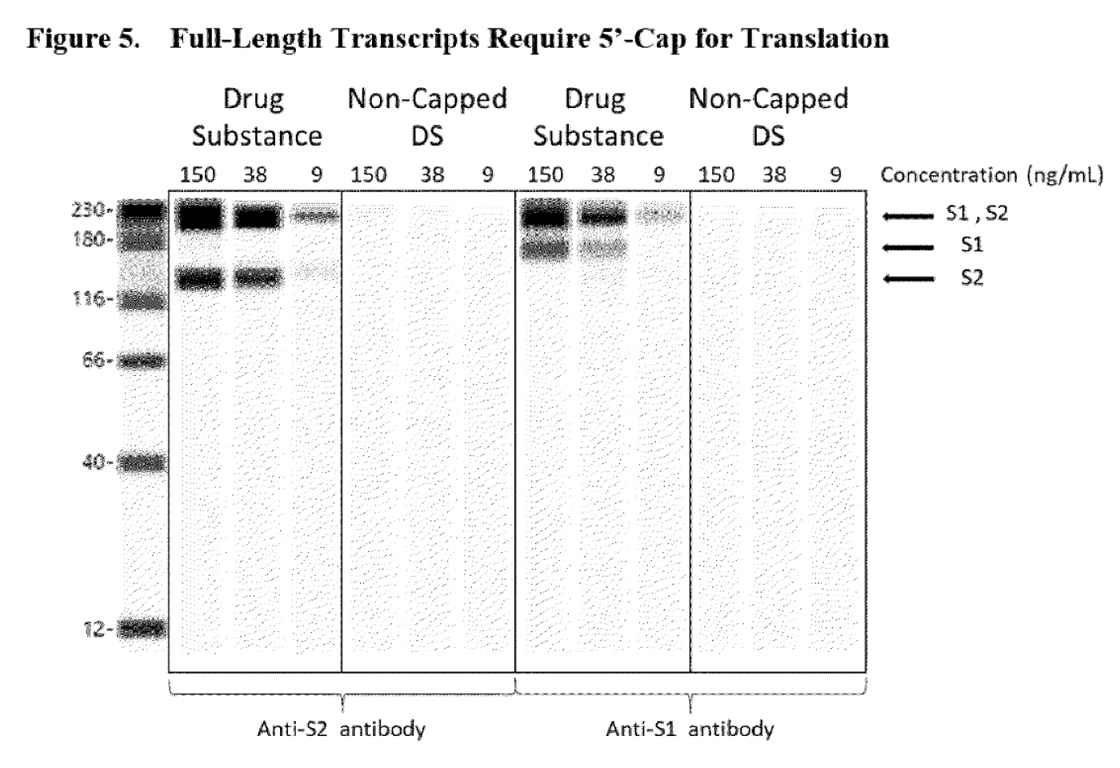
Además, se asumió que las especies de ARN fragmentadas eran el resultado de paradas transcripcionales prematuras o de la hidrólisis del ARNm (cuando una molécula se rompe en dos trozos al reaccionar con el agua), véase el extracto a continuación.
La evaluación de seguridad asociada reveló que la probabilidad de especies fragmentadas se genera por paradas transcripcionales prematuras o hidrólisis de ARNm. Como tal, las especies fragmentadas predominantemente no tienen tanto 5′-cap y poli (A) elementos de cola necesarios para la expresión de proteínas
Fuente: Informe del CHMP de la EMA de agosto de 2021
Esto dio una falsa sensación de seguridad de que un transcrito de ARNm truncado sólo podía tener una cola 5’cap o una cola poli (A), pero nunca ambas.
El espejismo del ensayo de degradación
BioNTech siguió investigando el potencial de los transcritos truncados de ARNm para producir proteínas a petición del regulador. Lo interesante es que el lote (1071509) fue seleccionado a propósito y degradado intencionadamente por exposición a temperatura elevada, con el fin de generar muestras con especies fragmentadas. El Western Blot que se muestra a continuación es el segundo tradicional presentado por BioNTech. Muestra que la capacidad del transcrito de ARN (cuando se degrada por el calor, y por tanto se fragmenta) para expresar una proteína es menor.
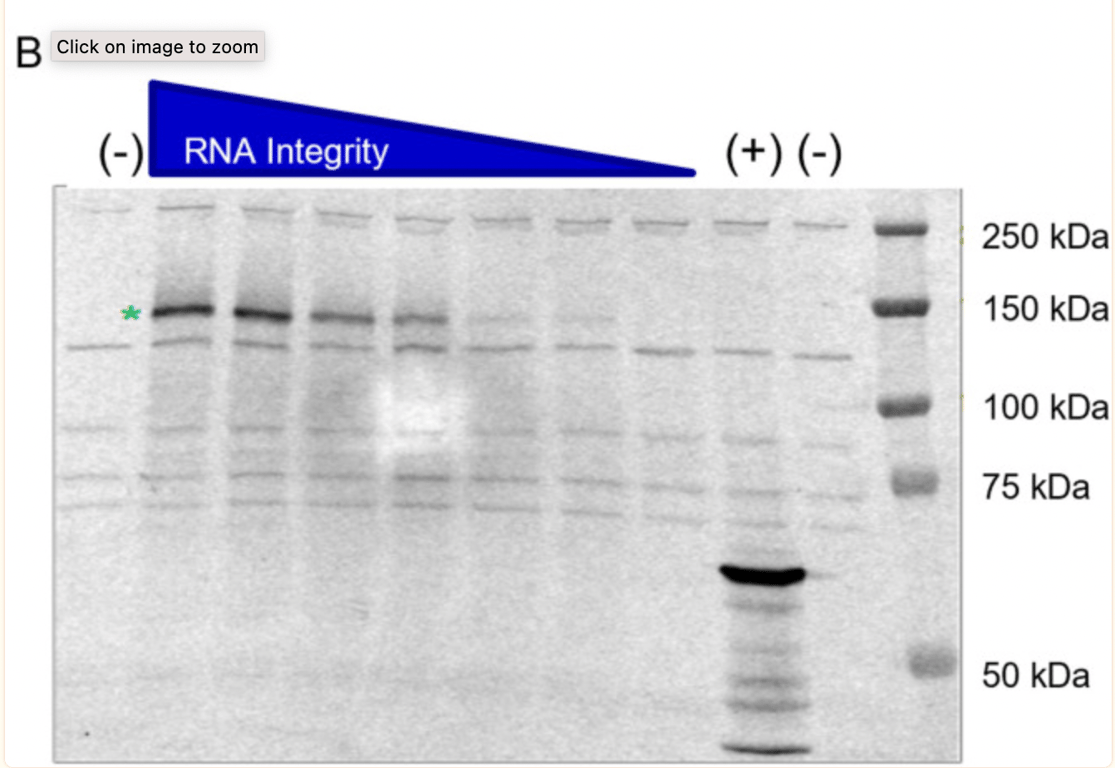
La estrella verde muestra que la muestra BNT162b2 no degradada (no aplicada calor, ARN intacto/alta integridad) produjo una proteína de aproximadamente 140kDa, consistente con el tamaño esperado de la proteína S1S2 aglicosilada. BioNTech afirma que «no se detectaron truncados ni otras especies de proteínas más allá de las bandas de fondo observadas en la muestra de control negativo», sin embargo, en el carril de la muestra no degradada, las bandas de proteínas son visibles y no se da ninguna explicación de lo que son.
En la entrevista de McKernon, explicó que «lo que hicieron en su estudio es que tomaron el ARNm y no lo fragmentaron de la forma que se obtiene en el proceso de fabricación, ¡sino que lo calentaron! La razón por la que están fragmentados es que las polimerasas se detienen en estas bases no nativas (modificadas) al sintetizar el ARN, por lo que se obtienen estos tramos más cortos de ARN que se hacen en el proceso de fabricación y ese proceso probablemente varía en función de los nucleótidos que obtienen de sus proveedores.»
En el informe de la EMA de agosto de 2021, se estableció una nueva obligación que BioNTech debía cumplir: una solicitud para que se realizara el mismo ejercicio de caracterización para al menos tres lotes adicionales de tozinameran (ARNm modificado, sustancia farmacológica). Como se puede ver, esto no se cumplió en agosto de 2021.

Y de acuerdo con la última actualización (2 Feb 2023) Comirnaty: Informe EPAR, esta obligación sigue sin cumplirse.

McKernan también hizo referencia al artículo de Patterson et al., que alarmantemente encontró versiones mutadas de la proteína de la espiga en individuos vacunados que no existían en los no vacunados que tenían COVID-19. Esto puede tomarse como datos observacionales del mundo real que muestran que las versiones mutadas (aberrantes) de la proteína espiga están siendo traducidas por el ARNm modificado por la vacuna.
En su comentario final, McKernan afirmó: «Los responsables de la reglamentación están dormidos al volante. Han abandonado el timón y dejan que esto vaya a velocidad de crucero»
No podría estar más de acuerdo.
Publicado originalmente en Trial Site News
Sugerir una corrección