Des preuves saisissantes suggèrent que BioNTech et Pfizer ont falsifié des données clés et d’autres scandales: Partie 2

Publié à l’origine sur Trial Site News
La partie 1 de mon rapport d’enquête publié dans Trial Site News s’est concentrée sur les « Western Blots » de Pfizer-BioNTech à l’aspect inhabituel – le scandale connu sous le nom de Blotgate. Les Western Blots ont été réalisés par BioNTech pour prouver la conformité de leur produit (aux autorités de réglementation), à savoir que seule la protéine spike attendue était exprimée in-vitro par l’ARNm modifié du vaccin et qu’elle était cohérente entre les différents lots.
Le fait que ces bandes ne ressemblent pas à des Western blots authentiques/conventionnels, mais plutôt à des versions générées par ordinateur (Western blots automatisés, bien qu’ils ne soient désignés comme tels dans aucun des rapports), ne fait que détourner l’attention du véritable scandale : les preuves, qui montrent, à première vue, que BioNTech/Pfizer ont « copié et collé » ces bandes, en d’autres termes, qu’ils ont falsifié leurs données clés. Ces bandes Western apparemment fabriquées (voir ci-dessous) ont été révélées dans la réponse de Pfizer aux questions de la Food and Drug Administration américaine vers novembre 2020, à l’approche de l’approbation de l’utilisation d’urgence.
Ce n’est que lorsque ces bandes ont été quantifiées à l’aide d’un logiciel d’analyse d’images (grâce à un expert anonyme) que le travail de « copier-coller », effectué sur 4 lots distincts de leur produit, transfecté à 6 concentrations différentes, est devenu remarquablement visible.
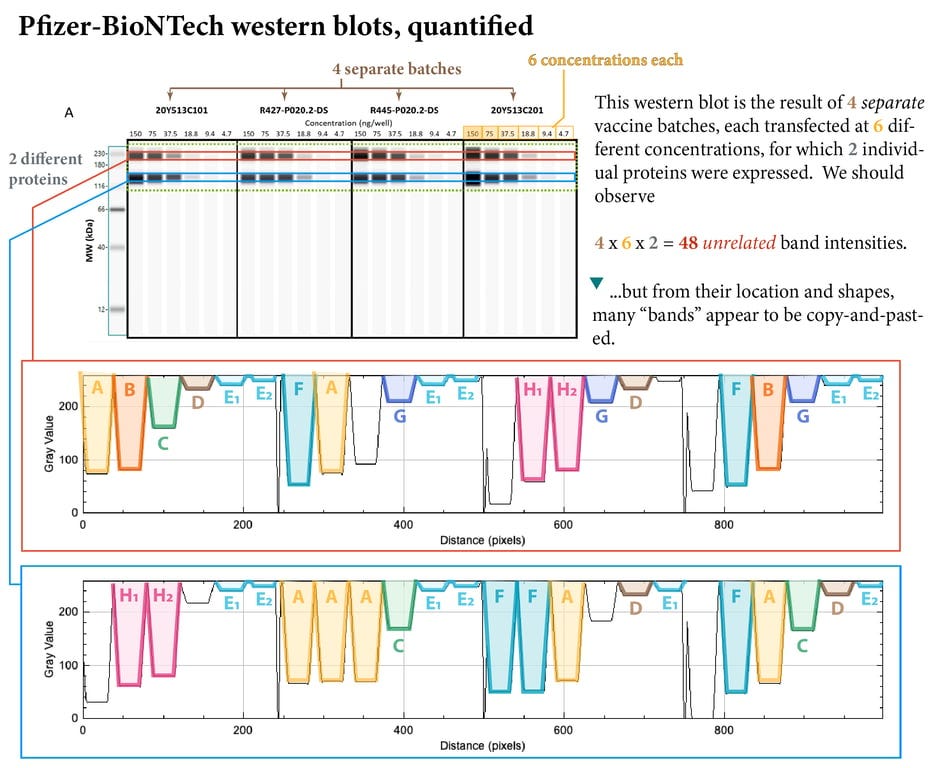
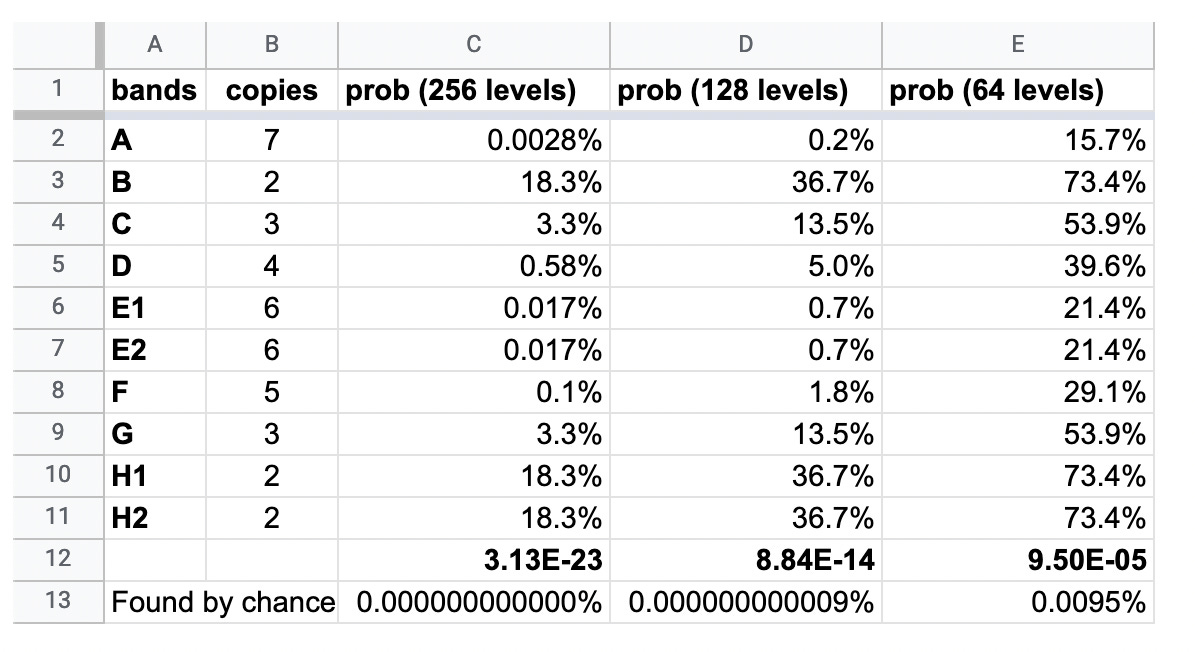
Les chances que les mêmes bandes apparaissent naturellement dans quatre lots distincts de vaccins, transfectés à 6 concentrations différentes, sont indiquées dans le tableau ci-dessous.
À la suite de la première partie de mon rapport d’enquête, Epoch Times a mené sa propre enquête sur le Blotgate, les problèmes de qualité de l’ARNm du vaccin Pfizer-BioNTech, et a fait référence à l’enquête de Trial Site News sur les fuites de courriels et de documents de l’Agence européenne des médicaments (EMA).
cacher les bandes sales » – un regard supplémentaire sur le Blotgate
Le 13 janvier, peu après l’éclatement du scandale du Blotgate, l’article sponsorisé par Pfizer et BioNTech, Patel et al. intitulé « Characterization of BNT162b2 mRNA to Evaluate Risk of Off-Target Antigen Translation » a été publié dans le Journal of Pharmaceutical Sciences. L’article présente de nombreuses anomalies et il convient de noter que le Dr Jessica Rose, biologiste moléculaire et informatique, a rédigé une analyse critique complète de cette étude.
Tout d’abord, il convient de noter les conflits d’intérêts des auteurs, que l’on peut voir ci-dessous.

Jessica Rose, qui a réalisé de nombreux Western Blots conventionnels, a expliqué : « Selon mon avis d’expert, [un Western traditionnel] est l’un des banc tests qui est une procédure, comme les tests de sécurité des produits biologiques (qui sont censés durer 10 ans). Un Western blot nécessite une séquence d’étapes spécifiques, ces étapes ne peuvent pas être précipitées et ne peuvent pas se chevaucher… les expériences réalisées à l’aide de Robo Jess [machine automatisée de Western blot] et les blots soumis par Pfizer doivent pouvoir être reproduits par des mains humaines, la reproductibilité est une exigence, en particulier lorsque la liste des conflits d’intérêts est si longue de la part des auteurs qui ont soumis l’étude [Patel et al.] »
L’image ci-dessous, tirée de l’article de Patel et al., montre leur « Western Blot ». On remarque les bandes noires régulières très épaisses (homogènes), sans aucune trainée.
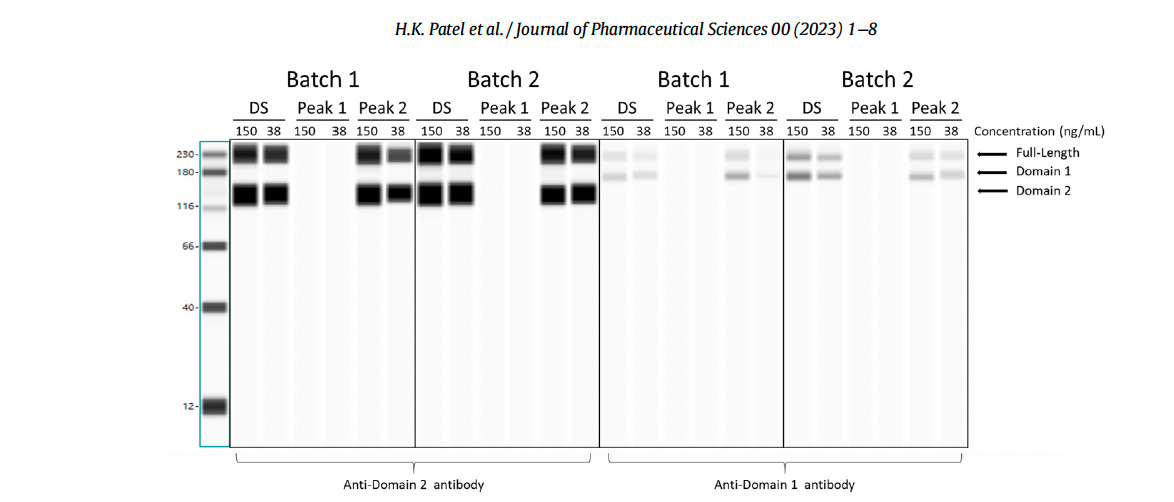
Le même « Western Blot » (voir ci-dessous), mais dans une version beaucoup plus grossière et scannée, se trouve dans le rapport expurgé du CHMP (Comité des médicaments à usage humain) de l’Agence européenne des médicaments d’août 2021, soit environ 18 mois avant l’article de Patel et al.
Étant donné que les auteurs Patel et al. font référence à la technologie ProteinSimple dans leur étude (extrait ci-dessous), une comparaison peut être faite avec un échantillon de Western blot automatisé utilisant le logiciel de la même société.
Des lysats cellulaires ont été analysés avec des anticorps spécifiques pour détecter les protéines Spike du SARS-CoV-2 à l’aide de la technologie ProteinSimple. Le module de séparation Wes 12-230 kDa et 25 cartouches capillaires ont été utilisés. Des anticorps de la sous-unité S1 du pic SARS-CoV-2 de souris (R&D systems, catalogue no. MAB105403) et des anticorps de la sous-unité S2 du pic SARS-CoV-2 de souris (R&D systems, catalogue no. MAB10557) ont été utilisés. Les résultats des échantillons d’ARNm sont présentés sous forme d’images du module de séparation Wes de ProteinSimple
Source : Patel et al
L’image ci-dessous représente un Western automatisé utilisant la technologie ProteinSimple.
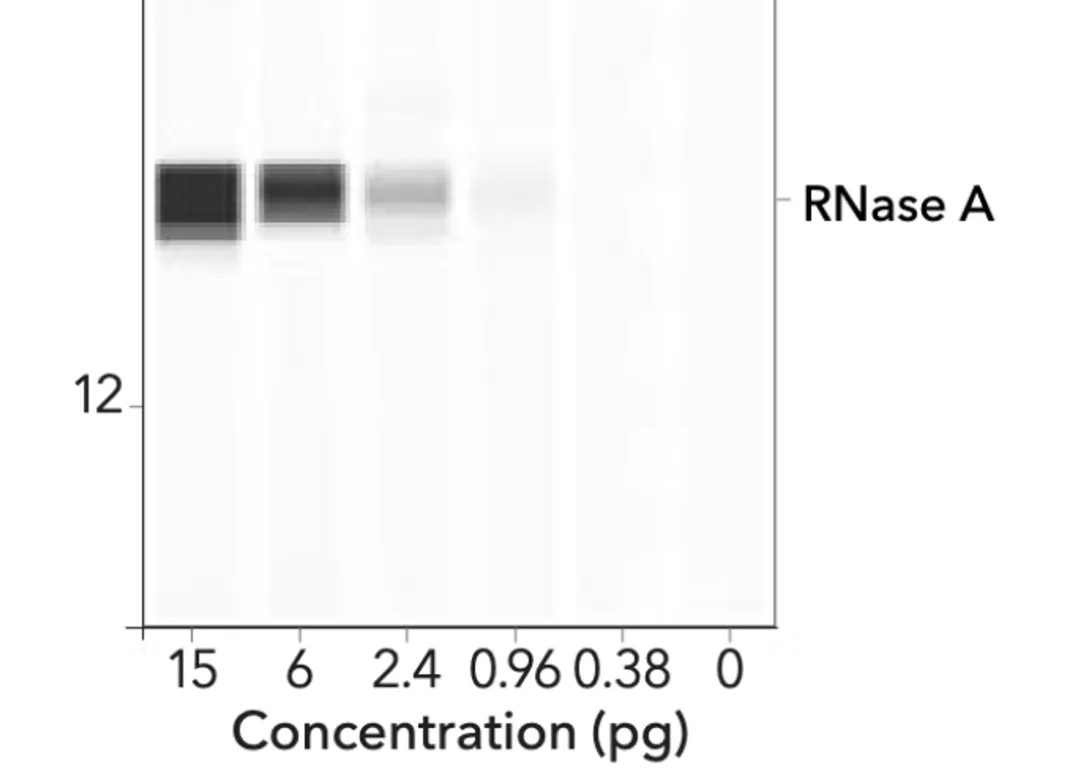
Ce qui est frappant, c’est qu’un gradient (effet d’étalement) peut être observé dans chacune des bandes variées. Ces bandes sont très différentes des bandes noires épaisses et homogènes de BioNTech/Pfizer.
Pour en savoir plus, nous avons interrogé Kevin McKernan, responsable de la R&D et scientifique en génomique du Human Genome Project, qui a expliqué que « les données de BioNTech/Pfizer sont inutilisables » et que l’aspect inhabituel des bandes de Western Blot (mentionné dans le rapport de l’EMA et l’article de Patel) s’explique par le fait que « BioNTech a considérablement augmenté le gain et que la plupart des gens font cela pour cacher les « bandes sales ». Il y a probablement 5 bandes ici [dans une bande épaisse qui semble unique] ». Je reviendrais sur cet entretien instructif plus loin dans le présent rapport.
Cela soulève la question suivante : BioNTech/Pfizer ont-ils dissimulé intentionnellement leurs résultats de « Western blot » en soumettant des versions manipulées (de Westerns automatisés) aux autorités de réglementation ? Peut-être s’agissait-il d’un travail de copier-coller pour la FDA et d’une manipulation des niveaux de saturation pour l’EMA ? Et plus important encore : comment les régulateurs ont-ils accepté ces « Western blots » comme preuve principale pour démontrer la conformité et la cohérence du produit de BioNTech et Pfizer ?
Nous avons contacté l’EMA pour obtenir une réponse aux questions soulevées dans cette enquête. Son attaché de presse a répondu par la déclaration suivante : « Ces figures [images de Western blot] ont été extraites du dossier soumis et insérées dans le rapport d’évaluation, ce qui a entraîné une perte de qualité de l’image. En outre, le logiciel de rédaction utilisé par l’EMA pour préparer les documents à publier à la suite d’une demande d’ATD a un impact sur la résolution des documents.
En ce qui concerne la validation des méthodes, les méthodes analytiques de Western Blot utilisées dans les études de caractérisation avaient déjà été évaluées lors de la demande initiale d’autorisation de mise sur le marché conditionnelle dans le cadre de l’historique du développement et de l’évaluation de la comparabilité » et ont été jugées appropriées à cette fin
La FDA, Pfizer et BioNTech n’ont pas répondu aux commentaires.
Paramètres de qualité établis plusieurs mois après l’octroi de l’autorisation de mise sur le marché
La raison pour laquelle le titulaire de l’autorisation de mise sur le marché (BioNTech) a effectué une série supplémentaire de tests est que l’autorisation de mise sur le marché conditionnelle (CMA) a été accordée sur la base du respect par le titulaire de l’autorisation de mise sur le marché d’obligations spécifiques (telles que des données supplémentaires pour caractériser davantage les espèces d’ARNm tronquées et modifiées) imposées par l’EMA. Au moment où l’autorisation de mise sur le marché a été accordée, plusieurs questions de CMC (chimie, fabrication et contrôle) avaient été soulevées comme des préoccupations majeures par l’EMA, en particulier la diminution de l’intégrité de l’ARNm (présence d’ARNm tronqué/fragmenté – dépourvu d’un attribut critique, d’une extrémité 5′ et/ou d’une queue poly(A)) des lots commerciaux par rapport à ceux utilisés dans les essais cliniques. La « solution » à ce problème majeur a été de réduire les critères d’acceptation des échantillons d’ARNm fragmentés/tronqués à 50 %, le seuil critique que les régulateurs ont tout simplement baissé.
La pharmacienne canadienne Maria Gutschi, PharmD, qui a plus de 30 ans d’expérience en milieu hospitalier, communautaire et gouvernemental, a examiné de manière indépendante les problèmes de qualité des vaccins Pfizer/BioNTech identifiés par l’Agence européenne des médicaments dans une présentation vidéo informative. Gutschi a soulevé d’autres préoccupations importantes : ce n’est qu’en mai 2021 [cinq mois après l’octroi de l’autorisation] que l’OCABR a établi les paramètres de qualité et, plus important encore, la normalisation des tests utilisés. Nous avons constaté qu’au moment du lancement, de nombreux tests utilisés pour déterminer la qualité et l’identité étaient des tests « internes » de BioNTech. Or, un organisme de réglementation ne peut pas accepter cela. Ils doivent être validés afin d’être cohérents, reproductibles et fiables
OCABR signifie Official Control Authority for Batch Release (Autorité officielle de contrôle pour la libération des lots), qui définit les lignes directrices pour les vaccins humains autorisés dans l’UE. Notez la date de mai 2021 mise en évidence dans le document de l’OCABR (voir ci-dessous).

Le fait que l’OCABR ait fixé les premiers paramètres de qualité pour un vaccin humain, cinq mois après l’octroi de l’autorisation de mise sur le marché, est sans précédent. Deuxièmement, il est intéressant de noter le fait que ces tests (essais) devaient être validés.
Des tests « internes » douteux
Il convient de noter que les préoccupations de Gutschi concernant les tests « internes » de BioNTech ont également été partagées par un examinateur de l’EMA dans le rapport d’examen continu du rapporteur de novembre 2020 qui a été divulgué peu avant l’octroi de l’AMC.

Si l’on se réfère au document guide de 2016 de la FDA sur l’intégrité des données et la conformité aux CGMP, on peut lire : « Intégrité des données et conformité aux CGMP » : « Ces dernières années, la FDA a observé de plus en plus d’infractions aux CGMP concernant l’intégrité des données lors d’inspections CGM. Cette situation est préoccupante car la garantie de l’intégrité des données est un élément important de la responsabilité de l’industrie à assurer la sécurité, l’efficacité et la qualité des médicaments et de la capacité de la FDA à protéger la santé publique »
Le fait que la FDA considère les violations de l’intégrité des données comme « préoccupantes » semble disparaître – en acceptant facilement les données de BioNTech qui semblent être des « copier-coller ». Il ne s’agissait peut-être pour eux que de cocher des cases pour donner l’impression qu’ils faisaient preuve de la diligence nécessaire pour « protéger la santé publique »
Bandes « inattendues » apparaissant dans les vrais Western blots de BioNTech
Parmi tous les « Western blots » générés par ordinateur, BioNTech a en fait soumis deux Westerns authentiques (le second sera discuté plus tard), ce qui prouve qu’ils savaient comment les faire. Dans le rapport d’évaluation continue de l’EMA qui a fait l’objet d’une fuite, les critiques suivantes ont été formulées à l’encontre de l’étude 20-0211 de BioNTech« en ce qui concerne les résultats des Western blots », comme indiqué ci-dessous.

En février dernier, l’étude 20-0211 (l’étude mentionnée ci-dessus) menée par BioNTech a été publiée dans le cadre de la publication des données de Pfizer ordonnée par le tribunal. Le vrai Western Blot montré dans cette étude, très révélateur, peut être vu ci-dessous.

D’après le rapport d’évaluation de l’EMA de novembre 2020, nous savons que l’agence a signalé les poids moléculaires inattendus (mesurés en kDa) des deux bandes de protéines montrées dans le Western Blot, 190 kDa et 100 kDa respectivement, ce qui l’a incitée à demander des explications à BioNTech. La protéine Spike complète qui a été utilisée comme contrôle a un poids moléculaire de 141 kDa, et la S1 (sous-unité de la protéine d’épi) a un poids moléculaire de 76,5 kDa. La figure ci-dessus montre qu’il n’y a pas de bandes de protéines à l’un de ces poids moléculaires attendus dans la bande BNT 162B2 et que le poids moléculaire attendu de 76,5 kDa de la protéine S1 n’a pas été observé dans la bande de contrôle S1. Ces anomalies ont attiré l’attention du régulateur car le développeur du vaccin, BioNTech, qui aurait dû connaître le poids moléculaire attendu de la protéine Spike complète, que leur produit ARNm était censé de coder, ne les a même pas reconnues.
Pour aggraver son incapacité à résoudre ces problèmes, BioNTech a rédigé une description extrêmement inexacte de son Western Blot – en fait, l’exact opposé de ce qui était montré sur l’image : « BNT162b2b2 a une taille attendue de 141,14 kDa » et « la protéine de la sous-unité S1 du SARS-CoV-2 (76,5 kDa) a été utilisée comme contrôle positif«
Cela suggère qu’il est probable que la protéine de pointe S1S2 n’ait pas été exprimée par l’ARNm modifié du vaccin ou, si elle l’a été, que d’autres protéines aberrantes provenant de molécules d’ARN fragmentées/tronquées dans la substance médicamenteuse aient également été exprimées.
Absence de séquençage du génome et problèmes liés à la méthylpseudourine N1
Interrogé sur les problèmes de qualité du vaccin Pfizer-BioNTech, le chercheur en génomique Kevin McKernan a donné la réponse suivante :
« Nous ne disposons d’aucun séquençage de l’ADN pour ces lots. C’est de la pure folie ! Dans le cadre du projet du génome humain, nous publions une séquence toutes les 24 heures pour nous assurer que le monde a accès aux données les plus récentes du projet du génome humain. Aujourd’hui, on ne trouve aucun séquençage de lots de génome«
Il est étonnant que les autorités réglementaires se soient appuyées sur ces tests Western blot d’apparence manipulée de BioNTech, au lieu d’exiger le séquençage du génome des lots pour prouver leur cohérence et leur conformité.
M. McKernan a ensuite abordé les problèmes causés par l’ARN modifié synthétisé à l’aide d’une base modifiée :« Nous avons un produit ARNm dans lequel chaque uridine a été remplacée par de la N1 méthylpseudo-uridine, ce que l’organisme n’a jamais vu auparavant. Ils [BioNTech et Pfizer] ont choisi des codons stop qui sont les plus connus pour créer des erreurs. Ils étaient conscients du problème mais ne l’ont pas résolu correctement. Cela signifie que lorsque les ribosomes lisent le modèle, ils sont très confus parce qu’ils ne l’ont jamais vu auparavant.
Peter McCullough et Anthony Kyriakopoulos, co-auteurs d’une publication intitulée « Differences in Vaccine and SARS-CoV-2 Replication Derived mRNA : Implications for Cell Biology and Future Disease » ont conclu que « les changements dans les codons synonymes intégrés dans les vaccins ARNm peuvent modifier la conformation attendue de la protéine codée, car la vitesse et l’efficacité de la traduction peuvent conduire à une conformation protéique différente… ». Les stratégies d’optimisation des codons pour le développement de vaccins à ARNm peuvent entraîner une dysrégulation immunitaire, affecter la régulation épi-transcriptomique et conduire à la progression de la maladie.
L’uridine modifiée (méthylpseudo-uridine N1) a été incorporée dans l’ARNm pour contourner la réponse immunitaire innée et favoriser la traduction des protéines. Cependant, dans le rapport de l’évaluation continue de l’EMA de novembre 2020 (page 61), un risque potentiel de sécurité avec l’ARN modifié (modRNA) a été soulevé, voir ci-dessous.
le MODRNA contient une substitution 1-méthyl-pseudo-uridine de l’uridine. Cette substitution diminue la reconnaissance de l’ARN vaccinal par les capteurs de l’immunité innée. Toutefois, aucune autre discussion sur le risque de réponses auto-immunes induites par le MODRNA n’a été fournie. Le demandeur est invité à discuter plus en détail de la possibilité que le vaccin ARNm puisse déclencher des réponses auto-immunes potentielles et de la manière dont il entend évaluer l’apparition éventuelle de telles réponses.
On ne sait pas si BioNTech a déjà évalué le risque potentiel de sécurité d’une réponse auto-immune causée par l’uridine modifiée ou les protéines traduites (autres que la protéine Spike), car à la date d’août 2021, l’échéance de juillet 2021 étant passée, cela n’a pas encore été fait. (Voir l’instantané ci-dessous, page 13 du rapport du CHMP de l’EMA d’août 2021)

Ce qui est inquiétant, ce sont les résultats de la plus grande étude de ce type menée par l’hôpital universitaire King Fahad de Khobar, en Arabie saoudite, qui établissent un lien entre les vaccins à ARNm et le déclenchement de maladies auto-immunes, ce que TrialSite l’a récemment rapporté. Le « mimétisme moléculaire », mentionné comme une préoccupation dans le rapport de l’EMA ci-dessus, est la même hypothèse que celle avancée dans l’étude comme mécanisme associé aux vaccins ARNm provoquant un processus auto-immun.
Problèmes potentiels liés au processus de fabrication
La figure ci-dessous illustre la vision simpliste utilisée pour promouvoir le fonctionnement des vaccins à ARNm.
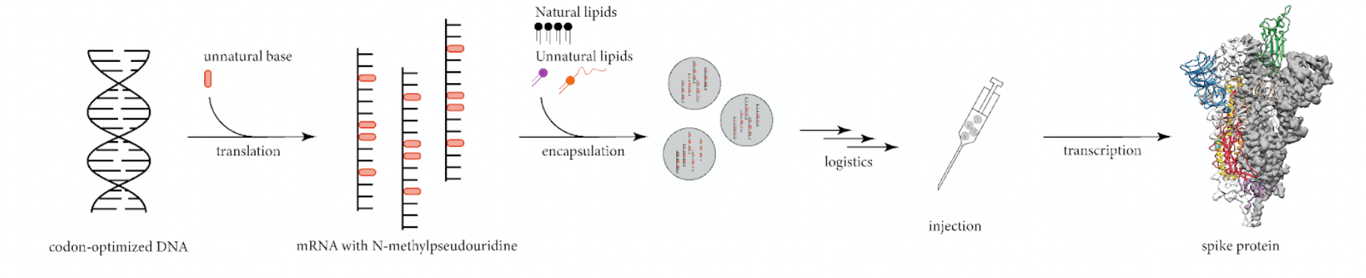
En réalité, le processus est très variable, ce qui génère à son tour des produits protéiques très variables (voir figure ci-dessous).

Chaque étape du processus de fabrication peut introduire des impuretés et des erreurs inconnues, depuis la traduction « infidèle » de l’ADN optimisé par codon, qui peut générer des fragments d’ARNm (non intacts), jusqu’à la transcription « infidèle » du mélange d’ARNm dans le corps humain, qui peut générer des produits protéiques inattendus.
McKernan a poursuivi en expliquant le taux d’erreur et ses implications : j’estime qu’il y a une erreur par molécule de vaccin – étant donné qu’il y a 14 billions de molécules dans chaque injection – et nous ne savons pas ce que cela signifie d’un point de vue immunologique. C’est pourquoi il faut procéder à un séquençage lot par lot avant d’injecter un produit dont on sait qu’il produirait de nouvelles protéines à l’intérieur du corps.
L’ARNm modifié complet du vaccin Pfizer-BioNTech codant pour la protéine Spike a une longueur de ∼4300 nucléotides (nt). Tout ce qui est inférieur est considéré comme une espèce d’ARNm fragmenté, que l’EMA a classé comme une « impureté liée au produit ». Cela conduit à un autre scandale, connu sous le nom de Humpgate.
Le scandale du Humpgate
Le Humpgate peut être considéré comme le précurseur du Blotgate. Alors que le Blotgate se concentrait principalement sur les bandes de protéines faussement exprimées de l’ARNm du vaccin observées dans les Westerns de BioNTech, le Humpgate fait référence aux espèces d’ARNm tronquées signalées par l’EMA et d’autres organismes de réglementation. Le nom vient des « bosses » que l’on voit dans l’image du message sur les médias sociaux ci-dessous.
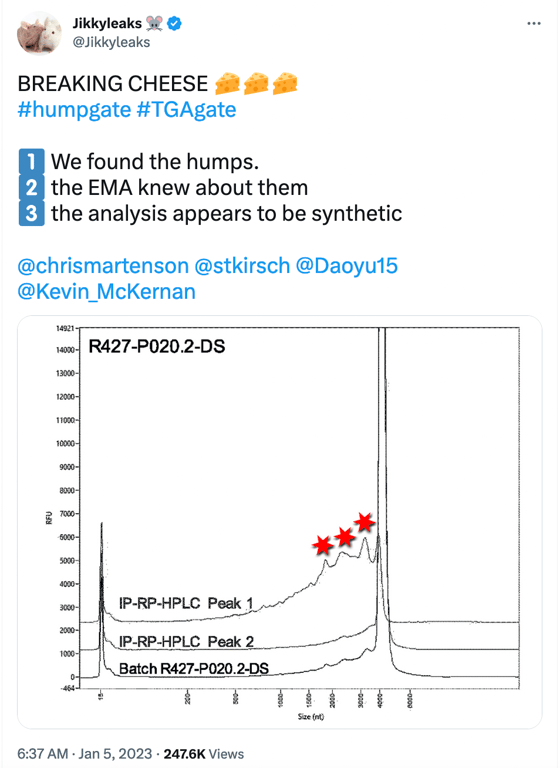
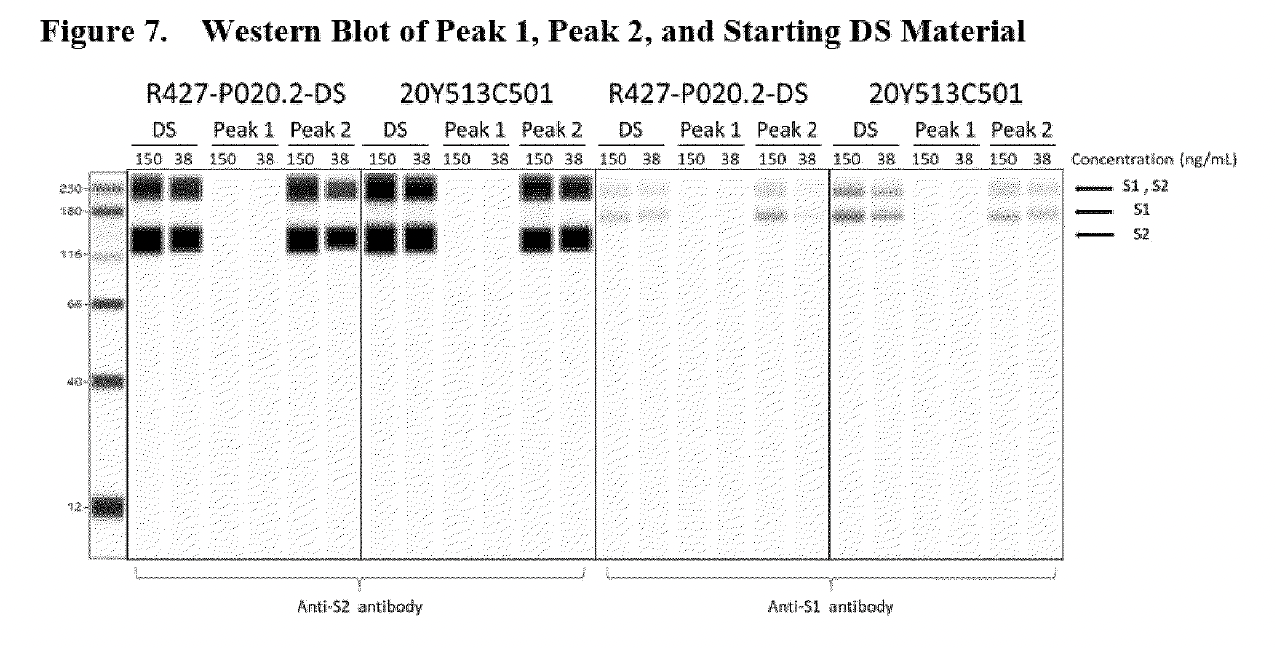
Les 3 « bosses » indiquées par les étoiles rouges représentent des espèces d’ARN tronquées/fragmentées ayant une longueur raccourcie (nt) <4000nt. L’image ci-dessus est un électrophérogramme de l’analyseur de fragments (qui est l’image du haut de la figure 2 ci-dessous) extrait de la page 15 du rapport de l’EMA rédigé en été 2021, que BioNTech a entrepris pour répondre à l’obligation spécifique 1 (SO1) demandé par l’EMA; celui ci demandait des données supplémentaires pour mieux caractériser les espèces d’ARNm tronquées et modifiées.

Des échantillons fractionnés ont été prélevés dans l’essai 1 (R427-P020.2-DS, lot d’essai clinique, image du haut) et l’essai 2 (20Y513C501, lot PPQ, image du bas) à l’aide de la RP-HPLC avec couplage d’ions pour mieux caractériser les espèces d’ARNm intactes (pleine longueur) et fragmentées. Les deux lots (essais 1 et 2) présentent de petites bosses conduisant à un pic important d’une taille d’environ 4 300 nt, qui représente l’espèce d’ARNm de pleine longueur. La ligne graphique étiquetée « Peak 1 » montre le matériel réanalysé, « purifié » et composé uniquement d’espèces d’ARNm fragmentaires (il s’agit des petites bosses observées sur le côté gauche du grand pic d’environ 4300 nt dans l’échantillon du lot), et « Peak 2 » est le matériel réanalysé, « purifié » et composé d’espèces d’ARNm intactes observées au niveau du « grand pic ».
Le même électrophérogramme se trouve dans l’article publié en janvier 2023 par Patel et al. (voir l’image ci-dessous, publiée 18 mois après le rapport de l’EMA). Cependant, les auteurs Patel et al. citent leur recherche comme étant actuelle, alors qu’il s’agit d’une version retravaillée de la réponse de Pfizer/BioNTech aux questions de la FDA et de l’EMA datant d’il y a 18 mois. Le fait que les auteurs présentent leur étude comme « indépendante » et « actuelle » alors que la technologie ProteinSimple Wes, utilisée dans leur recherche n’était plus utilisée depuis le 30 juillet 2021 est pour le moins fallacieux.
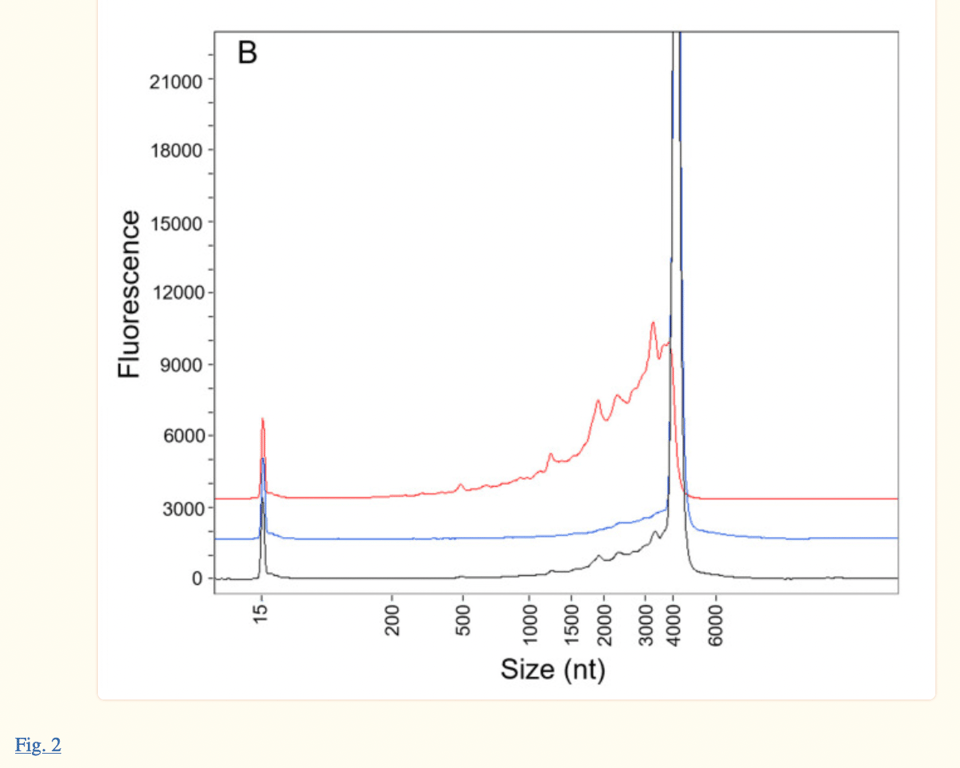
L’article de Patel et al. et l’article de l’EMA discutent tous deux des bosses observées sur le côté gauche du pic principal (à environ 4300 nt). Le rapport de l’EMA indique que « (la figure 2) démontre que le pic 1 est presque entièrement constitué d’espèces fragmentées, ce qui est cohérent avec les données précédemment fournies dans l’évaluation de la réponse à la Q01-Qualité du CHMP 11-Déc-2020 »
Le problème avec l’hypothèse de BioNTech
La façon dont BioNTech a assuré à l’EMA que ces espèces d’ARN tronquées ne soutiendraient pas la traduction des protéines, de sorte que les espèces tronquées, vues comme des « bosses » dans les électrophérogrammes, ne seraient pas considérées comme un problème, a été de réaliser de nouveaux essais Western Blot pour prouver que « les transcrits de l’ARN ont besoin à la fois de 5′-cap et de poly(A) pour soutenir la traduction des protéines… »

Voici à quoi ressemble un ARN BNT162b2 de pleine longueur. Une coiffe de 5′ est ajoutée au début de la transcription de l’ARN et une queue de 3′ poly(A) est ajoutée à la fin de la transcription. Les ribosomes lisent la transcription dans la direction du 5′ à 3′.

Les figures ci-dessous (6 et 5) sont extraites du rapport CHMP de l’EMA d’août 2021.

Les lignes blanches figurant dans les panels de substances médicamenteuses non poly(A) étaient suffisantes pour prouver à l’autorité réglementaire que l’ARN dépourvu de poly(A) ne serait pas en mesure d’exprimer la protéine de pointe S1S2. Toutefois, le problème de ce test est qu’il ne confirme pas que d’autres protéines aberrantes (ce qui était une préoccupation initiale de l’EMA) pourraient être exprimées par cet ARN tronqué, car seuls des anticorps de détection spécifiques des domaines S1 et S2 ont été utilisés. Le Western blot présenté ci-dessous présente le même problème. Ce test a été utilisé pour rassurer l’autorité de régulation sur le fait que le transcrit d’ARN dépourvu de 5’cap ne pouvait pas exprimer la protéine de pointe S1S2.
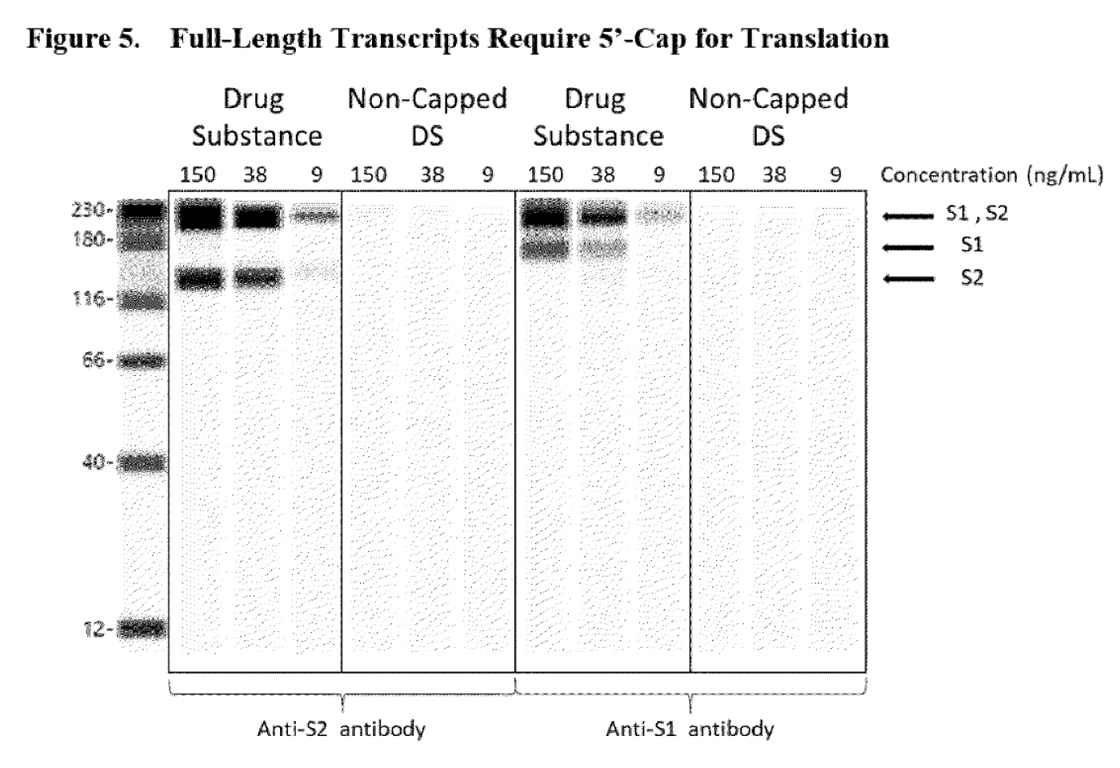
En outre, il a été supposé que l’espèce d’ARN fragmenté était le résultat d’arrêts prématurés de la transcription ou de l’hydrolyse de l’ARNm (lorsqu’une molécule se brise en deux morceaux en réagissant avec de l’eau), voir l’extrait ci-dessous.
l’évaluation de la sécurité associée a montré que la probabilité d’espèces fragmentées était générée par des arrêts prématurés de la transcription ou par l’hydrolyse de l’ARNm. En tant que telles, les espèces fragmentées sont principalement dépourvues des éléments 5′-cap et poly(A) tail nécessaires à l’expression de la protéine
Source : Rapport CHMP de l’EMA d’août 2021
Cela a donné un faux sentiment de sécurité selon lequel un transcrit d’ARNm tronqué pourrait seulement avoir une coiffe 5′- ou une queue poly(A), mais jamais les deux.
Le mirage du test de dégradation
Le potentiel des transcrits d’ARN tronqués à produire des protéines a été étudié plus avant par BioNTech à la demande de l’organisme de réglementation. Ce qui est intéressant, c’est que le lot (1071509) a été intentionnellement sélectionné et intentionnellement dégradé par exposition à des températures élevées pour générer des échantillons avec des espèces fragmentées. Le Western blot présenté ci-dessous est le deuxième traditionnel Western Blot soumis par BioNTech. Il montre que la capacité de l’ARN transcrit (lorsqu’il est dégradé par la chaleur, donc fragmenté) à exprimer une protéine devient plus faible.
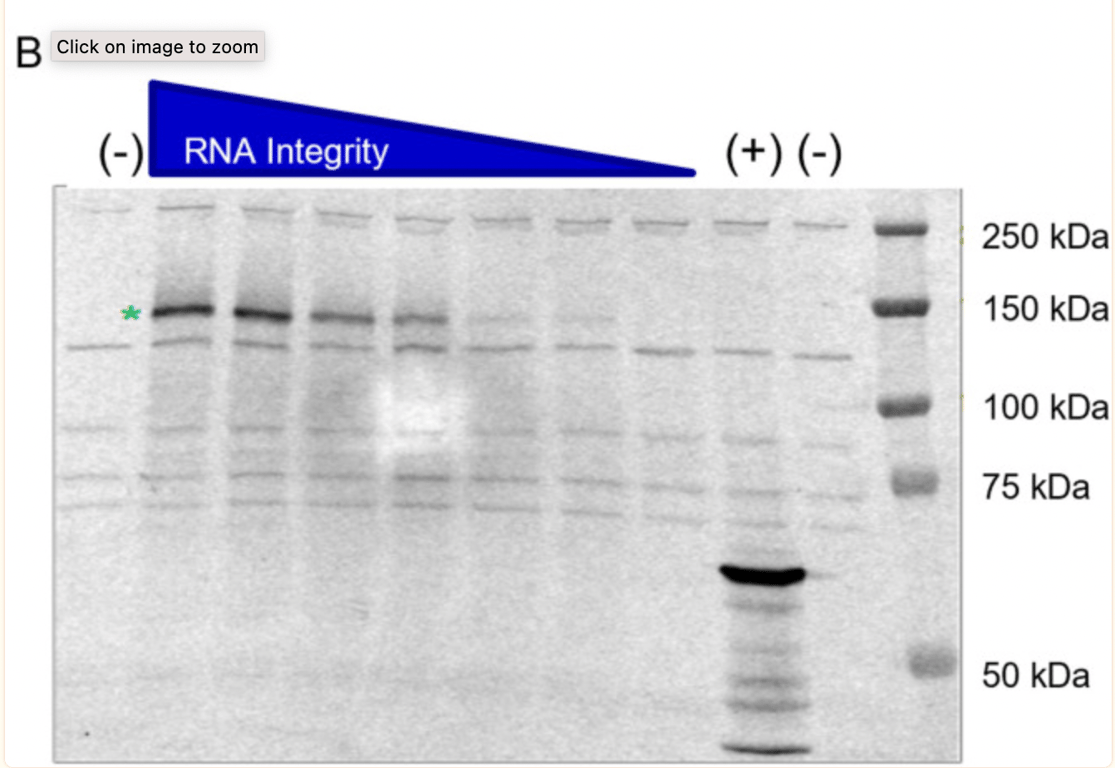
L’étoile verte montre que l’échantillon BNT162b2 non dégradé par la chaleur (pas de chaleur, ARN intact/à haute intégrité) a produit une protéine d’environ 140 kDa, ce qui correspond à la taille attendue de la protéine S1S2 aglycosylée. BioNTech déclare qu’ « aucune espèce de protéine tronquée ou autre n’a été détectée au-delà des bandes de fond observées dans l’échantillon de contrôle négatif », cependant, dans la bande de l’échantillon non dégradé, des bandes de protéines sont visibles et aucune explication n’est donnée quant à leur nature.
Dans l’interview de McKernon, il explique : « Ce qu’ils ont fait dans leur étude, c’est qu’ils ont pris de l’ARNm et qu’ils ne l’ont pas fragmenté comme on le fait dans le processus de fabrication, ils l’ont chauffé ! La raison pour laquelle ils sont fragmentés est que les polymérases se bloquent sur ces bases non natives (modifiées) dans la synthèse de l’ARN, de sorte que vous obtenez ces portions plus courtes d’ARN dans le processus de fabrication, et ce processus varie probablement en fonction des nucléotides qu’ils obtiennent de leurs fournisseurs »
Dans le rapport de l’EMA d’août 2021, une nouvelle exigence a été formulée à l’égard de BioNTech – une demande pour que le même exercice de caractérisation soit effectué pour au moins trois lots supplémentaires de tozinameran (ARNm modifié, substance médicamenteuse). Comme vous pouvez le constater, cet exigence n’a pas été répondue en août 2021.

Et selon la dernière mise à jour (2 février 2023) du rapport Comirnaty : Rapport EPAR, cette obligation n’a toujours pas été remplie.

McKernan a également fait référence aux travaux de Patterson et al. qui, de manière alarmante, ont trouvé des versions mutantes de la protéine Spike chez les personnes vaccinées qui n’existaient pas chez les personnes non vaccinées atteintes de COVID-19. Cela peut être considéré comme des données d’observation du monde réel montrant que les versions mutantes (aberrantes) de la protéine Spike sont traduites par l’ARNm modifié par le vaccin.
Dans son commentaire final, M. McKernan a déclaré : « Les responsables de la réglementation dorment au volant. Ils ont quitté le gouvernail et ont laissé le régulateur de vitesse fonctionner »
Je suis tout à fait d’accord.
Publié à l’origine sur Trial Site News
Suggérer une correction